Выявление различных структур в генетических данных различного уровня и различного таксономического и функционального положения, а также исследование связи таких структур между собой и с другими биологически важными показателями является ключевой проблемой современной молекулярной биологии и биоинформатики. В данной работе рассмотрена связь между статистическими особенностями сравнительно коротких фрагментов геномов хлоропластов и теми структурами в пространстве частот триплетов, определяемых по этим фрагментам (кластеров), которые они формируют. Выбор таких генетических объектов обусловлен, прежде всего, тем, что у них одинаковая функция. Во-вторых, геномы хлоропластов имеют сравнительно малый размер (105 0п.н.). В-третьих, происхождение хлоропластов традиционно ведут от бактерий [3]. Целью настоящей работы является выявление характерных особенностей геномов хлоропластов.
Ранее [1, 2] было показано, что геномы бактерий обладают удивительной семикластерной структурой: если геном бактерии формально разбить на набор (пересекающихся) участков одинаковой длины (порядка ≈102 нуклеотидов), затем для каждого из них построить частотный словарь толщины 3 и полученные словари кластеризовать в полученном пространстве частот триплетов, то все выделенные фрагменты разойдутся по семи кластерам. Данная работа посвящена проверке этого свойства для геномов хлоропластов различных наземных многоклеточных растений. Анонсируя основной результат работы, сразу скажем, что наши наблюдения показывают заметные различия между картиной семикластерного распределения у бактерий и у хлоропластов.
Введём основные понятия. Частотный словарь толщины 3 символьной последовательности, соответствующей ДНК — список всех троек 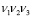 идущих подряд нуклеотидов с указанием их частот; всего бывает 64 триплета. Частота — это отношение числа копий
идущих подряд нуклеотидов с указанием их частот; всего бывает 64 триплета. Частота — это отношение числа копий 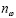 данного слова к общему числу всех триплетов:
данного слова к общему числу всех триплетов:
(1)
Всякий частотный словарь отображает геном в 64-мерное метрическое пространство; близость двух геномов задается естественным образом — например, как близость двух точек в Евклидовой метрике:
(2)
Один из 64 триплетов исключался, поскольку сумма всех частот в словаре равна 1 — наличествует линейная связь, которая будет давать ложный сигнал при статистической обработке (корреляционном анализе, определении главных компонент и т.п.). Формально исключить можно любой триплет, однако есть несколько эвристических правил для исключения. Первое правило состоит в том, чтобы исключить самый большой по значению частоты триплет, особенно, если значение его частоты на порядок (или около того) превосходит значение частоты следующего за ним (по этой величине) триплета. Второй подход состоит в том, чтобы исключать тот триплет, для которого стандартное отклонение, наблюдаемое по анализируемому набору фрагментов геномов, является минимальным: такой триплет даёт наименьший вклад в различимость объектов (в предельном случае, когда стандартное отклонение равно 0, различий вовсе нет). В наших исследованиях в основном исключались триплеты  и
и  , хотя ими список не исчерпывается.
, хотя ими список не исчерпывается.
Для выявления структурной близости необходимо выделить в этом 63-мерном метрическом пространстве группы точек (геномов), которые образуют чётко выявляемые кластеры. Каждый фрагмент нумеровался в зависимости от того, входил он в состав кодирующих или некодирующих участков. Если фрагмент входил в состав некодирующего участка, ему присваивалось число 4. Если фрагмент входил в кодирующий участок, то вычислялась его относительная фаза: остаток от деления номера в генетической последовательности первого нуклеотида в рассматриваемом фрагменте на номер в генетической последовательности первого нуклеотида в соответствующем кодирующем участке. Кроме того, учитывалось направление транскрипции генов. Таким образом, фрагментам, входящим в кодирующие участки, ставились в соответствие числа 0, 1 и 2 для прямого направления и 10, 11 и 12 для обратного направления.
Материалы и методы
Данные для исследования брались на сайте www.ebi.ac.uk/genomes/organelles в базе EMBL–банка. Были отобраны 188 хлоропластов наземных растений. По полученному частотному словарю в программе VidaExpert (http://bioinfo-out.curie.fr/projects/vidaexpert/) строился вид данных в пространстве первых трех главных компонент. Рассматривались две проекции пространства главных компонент, если можно так сказать, вид данных «анфас» и «в профиль». Точки данных были раскрашены в соответствии с их принадлежностью к не кодирующим участкам, либо в соответствии с относительной фазой для кодирующих участков. Точки, соответствующие не кодирующим участкам, окрашивались в коричневый цвет, точки, соответствующие относительным фазам 0 и 10, окрашивались в темно сиреневый и светло сиреневый цвет, точки, соответствующие относительным фазам 1 и 11, окрашивались в светло зеленый и темно зеленый цвет, точки, соответствующие относительным фазам 2 и 12, окрашивались в светло желтый и темно желтый цвет. Кроме того, для каждого генома вычислялся CG-контент, то есть отношение суммарного числа нуклеотидов C и G к общему числу нуклеотидов в геноме.
Результаты исследования и их обсуждение
Было обнаружено, что все геномы «анфас» имеют выраженную трех лучевую структуру. Для подавляющего большинства геномов типичен вид «анфас» и «в профиль», который показан на рис. 1.
Как видно на рис. 1, вид «анфас» хорошо кластеризован относительно точек из кодирующих и не кодирующих участков генома. Центральную часть рисунка «анфас» занимают точки из некодирующих участков, они окрашены в коричневый цвет. Один луч состоит из точек, относящихся к прямой и обратной относительной фазе 2 (точки, отмеченные светло желтым и темно желтым цветом), второй луч содержит точки, относящиеся к прямой относительной фазе 0 и обратной относительной фазе 1 (точки темно сиреневого и светло зеленого цвета), и третий состоит из точек, относящихся к обратной относительной фазе 0 и прямой относительной фазе 1 (точки светло сиреневого и темно зеленого цвета). На проекции «в профиль» видно, что точки, относящиеся к не кодирующим участкам, разделены на два кластера. Первый находится в центре трех лучевой структуры, а второй обособлен от остальных данных. Типичной структурой данных в пространстве первых трех главных компонент обладают геномы 170 хлоропластов из 188 рассматриваемых.
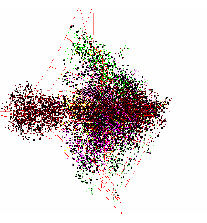
У оставшихся геномов хлоропластов наблюдались следующие отличия от типичной структуры данных. У геномов хлоропластов Selaginella moellendorffii и Selaginella uncinata отсутствует обособленная группа точек, относящаяся к некодирующим участкам генома. Это представители древних споровых растений, которые впервые появились около 400 млн. лет назад. Структура данных этих геномов существенно больше похожа на структуру данных геномов бактерий (см. Рис.2).
Геном хлоропласта Anthoceros angustus отличается от структуры, характерной для подавляющего большинства геномов хлоропластов: здесь обособленный участок данных этого хлоропласта состоит из точек кодирующих участков (рис. 3). Это единственный представитель Anthoceros в рассматриваемой базе генетических данных.
У растений Brachypodium distachyon, Juniperus bermudiana, Panax ginseng, Trachelium caeruleum, Fagopyrum esculentum, Pinus contorta, Syntrichia ruralis, Ptilidium pulcherrimum, Jacobaea vulgaris, Gossypium anomalum, Najas flexilis, Lupinus luteus, Glycyrrhiza glabra, Primula poissonii, Paeonia obovate относительные фазы в лучах перемешаны (см. рис. 4).
Для всех геномов хлоропластов вычислялся CG-контент, то есть отношение суммарного количества нуклеотидов C и G к общему количеству нуклеотидов в генетической последовательности. Обнаружилось, что для мхов CG-контент меньше 0.3 и изменялся от 0.28361 у Orthotrichum rogeri до 0.293164 у Sanionia uncinata. Для папоротников CG-контент больше 0.4 и изменялся от 0.406422 у Lygodium japonicum до 0.427245 у Myriopteris lindheimeri. У Selaginella moellendorffii и Selaginella uncinata, представителей древних споровых высших растений, CG-контент составил 0.510026 и 0.548491, соответственно. Для всех остальных геномов хлоропластов CG-контент изменялся от 0.3 до 0.4.
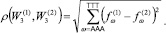
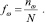
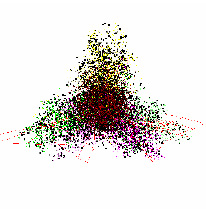
«анфас»
Рис. 1. Типичный вид структуры данных геномов наземных растений в проекциях пространства первых трех главных компонент (приведена структура генома Nimphaea alba)
«в профиль»
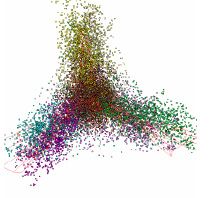
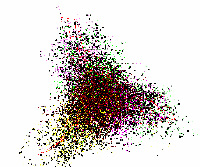
Рис. 2. Структура данных в проекциях пространства трех первых главных компонент Selaginella moellendorffii
Рис. 3. Структура данных в проекциях пространства трех первых главных компонент Anthoceros angustus
Рис. 4. Структура данных со смешанными фазами в лучах (Lupinus luteus)
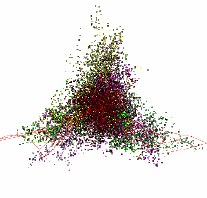

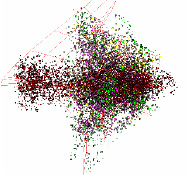

Таблица 1
CG-контент геномов хлоропластов
|
Название |
CG |
Название |
CG |
Название |
CG |
|
Orthotrichum rogeri |
0,284 |
Syntrichia ruralis |
0,284 |
Physcomitrella patens |
0,285 |
|
Physcomitrella patens |
0,285 |
Physcomitrella patens |
0,285 |
Marchantia polymorpha |
0,288 |
|
Sanionia uncinata |
0,293 |
Anthoceros angustus |
0,329 |
Ptilidium pulcherrimum |
0,332 |
|
Equisetum arvense |
0,334 |
Glycyrrhiza glabra |
0,342 |
Trifolium subterraneum |
0,344 |
|
Orobanche gracilis |
0,346 |
Taxus mairei |
0,346 |
Millettia pinnata |
0,348 |
|
Pisum sativum |
0,348 |
Juniperus virginiana |
0,348 |
Juniperus bermudiana |
0,349 |
|
Juniperus monosperma |
0,349 |
Lathyrus sativus |
0,351 |
Vigna radiata |
0,351 |
|
Jatropha curcas |
0,352 |
Glycine max |
0,354 |
Paphiopedilum armeniacum |
0,354 |
|
Angiopteris evecta |
0,354 |
Spirodela polyrhiza |
0,357 |
Lemna minor |
0,357 |
|
Wolffia australiana |
0,359 |
Manihot esculenta |
0,359 |
Pellia endiviifolia |
0,359 |
|
Apopellia endiviifolia |
0,359 |
Lotus japonicus |
0,360 |
Psilotum nudum |
0,360 |
|
Psilotum nudum |
0,360 |
Parinari campestris |
0,362 |
Huperzia lucidula |
0,362 |
|
Raphanus sativus |
0,363 |
Arabidopsis thaliana |
0,363 |
Arabidopsis thaliana |
0,363 |
|
Silene chalcedonica |
0,363 |
Nasturtium officinale |
0,364 |
Morus indica |
0,364 |
|
Pachycladon cheesemanii |
0,364 |
Lepidium virginicum |
0,365 |
Olimarabidopsis pumila |
0,365 |
|
Lobularia maritima |
0,365 |
Habenaria pantlingiana |
0,366 |
Typha latifolia |
0,366 |
|
Lupinus luteus |
0,366 |
Phalaenopsis equestris |
0,367 |
Welwitschia mirabilis |
0,367 |
|
Populus alba |
0,367 |
Populus alba |
0,367 |
Prunus kansuensis |
0,368 |
|
Pentactina rupicola |
0,368 |
Vaccinium macrocarpon |
0,368 |
Allium cepa |
0,368 |
|
Masdevallia coccinea |
0,368 |
Hibiscus syriacus |
0,368 |
Stockwellia quadrifida |
0,369 |
|
Quercus aliena |
0,369 |
Fritillaria hupehensis |
0,370 |
Trigonobalanus doichangensis |
0,370 |
|
Salix interior |
0,370 |
Primula poissonii |
0,370 |
Podocarpus lambertii |
0,371 |
|
Rosa odorata |
0,372 |
Fragaria chiloensis |
0,372 |
Potentilla micrantha |
0,372 |
|
Phoenix dactylifera |
0,372 |
Retrophyllum piresii |
0,373 |
Nageia nagi |
0,373 |
|
Nicotiana sylvestris |
0,373 |
Goodyera fumata |
0,373 |
Penthorum chinense |
0,373 |
|
Hordeum vulgare |
0,373 |
Jacobaea vulgaris |
0,373 |
Gossypium anomalum |
0,373 |
|
Triticum aestivum |
0,373 |
Vitis rotundifolia |
0,374 |
Vitis vinifera |
0,374 |
|
Lactuca sativa |
0,375 |
Utricularia gibba |
0,376 |
Hyoscyamus niger |
0,376 |
|
Ipomoea batatas |
0,376 |
Parthenium argentatum |
0,376 |
Helianthus annuus |
0,376 |
|
Guizotia abyssinica |
0,376 |
Rhazya stricta |
0,377 |
Viviania marifolia |
0,377 |
|
Iochroma nitidum |
0,377 |
Veratrum patulum |
0,377 |
Sedum sarmentosum |
0,377 |
|
Название |
CG |
Название |
CG |
Название |
CG |
|
Lindenbergia philippensis |
0,378 |
Olea europaea |
0,378 |
Schefflera delavayi |
0,378 |
|
Solanum lycopersicum |
0,379 |
Premna microphylla |
0,379 |
Ranunculus macranthus |
0,379 |
|
Metapanax delavayi |
0,379 |
Kalopanax septemlobus |
0,379 |
Liquidambar formosana |
0,379 |
|
Mankyua chejuensis |
0,380 |
Fagopyrum esculentum |
0,380 |
Jasminum nudiflorum |
0,380 |
|
Nelumbo lutea |
0,380 |
Trochodendron aralioides |
0,380 |
Salvia miltiorrhiza |
0,380 |
|
Megaleranthis saniculifolia |
0,380 |
Schwalbea americana |
0,381 |
Panax ginseng |
0,381 |
|
Tetracentron sinense |
0,381 |
Gnetum montanum |
0,382 |
Gnetum montanum |
0,382 |
|
Pinguicula ehlersiae |
0,382 |
Sesamum indicum |
0,382 |
Najas flexilis |
0,382 |
|
Lolium perenne |
0,382 |
Triticum aestivum |
0,383 |
Piper cenocladum |
0,383 |
|
Triticum aestivum |
0,383 |
Triticum aestivum |
0,383 |
Trachelium caeruleum |
0,383 |
|
Genlisea margaretae |
0,383 |
Neyraudia reynaudiana |
0,384 |
Pharus lappulaceus |
0,384 |
|
Paeonia obovata |
0,384 |
Saccharum hybrid |
0,384 |
Zea mays |
0,384 |
|
Pinus contorta |
0,384 |
Sorghum bicolor |
0,385 |
Pinus taeda |
0,385 |
|
Keteleeria davidiana |
0,386 |
Brachypodium distachyon |
0,386 |
Lonicera japonica |
0,386 |
|
Panicum virgatum |
0,386 |
Setaria italica |
0,386 |
Lecomtella madagascariensis |
0,386 |
|
Phragmites australis |
0,387 |
Picea abies |
0,387 |
Pseudotsuga sinensis |
0,388 |
|
Larix decidua |
0,388 |
Thamnocalamus spathiflorus |
0,388 |
Phyllostachys propinqua |
0,389 |
|
Phyllostachys edulis |
0,389 |
Hypseocharis bilobata |
0,389 |
Sarocalamus faberi |
0,389 |
|
Fargesia nitida |
0,389 |
Yushania levigata |
0,389 |
Indosasa sinica |
0,389 |
|
Pleioblastus maculatus |
0,389 |
Gaoligongshania megalothyrsa |
0,389 |
Oligostachyum shiuyingianum |
0,389 |
|
Puelia olyriformis |
0,390 |
Oryza sativa Japonica |
0,390 |
Illicium oligandrum |
0,390 |
|
Nuphar advena |
0,391 |
Oenothera argillicola |
0,391 |
Welwitschia mirabilis |
0,391 |
|
Nymphaea alba |
0,392 |
Liriodendron tulipifera |
0,392 |
Magnolia kwangsiensis |
0,393 |
|
Cycas revoluta |
0,394 |
Stangeria eriopus |
0,395 |
Ginkgo biloba |
0,396 |
|
Pelargonium x hortorum |
0,396 |
Zamia furfuracea |
0,397 |
Aneura mirabilis |
0,406 |
|
Lygodium japonicum |
0,406 |
Pteridium aquilinum |
0,415 |
Ophioglossum californicum |
0,422 |
|
Marsilea crenata |
0,422 |
Myriopteris lindheimeri |
0,427 |
Selaginella moellendorffii |
0,510 |
|
Selaginella uncinata |
0,548 |
(продолжени табл.1)
Заключение
Для большинства рассмотренных геномов хлоропластов наземных растений установлено, что структура данных в пространстве первых трех главных компонент очень похожа: имеется центральное ядро из фрагментов некодирующих участков и три луча, состоящих из фрагментов кодирующих участков. Кроме того, имеется отдельная группа фрагментов некодирующих участков, не входящая в центральное ядро. Следует заметить, что геномы хлоропластов обладают заметными отличиями по своей структуре от геномов бактерий: у них выделяется группа фрагментов (как правило, попадающих в некодирующие области), которые в пространстве главных компонент выглядят как своего рода «хвост». Этой особенности не наблюдается у геномов бактерий. Проведённые исследования показывают, что в отличие от бактериальных геномов, CG-контент не является здесь ведущим фактором распределения на типы геномов по числу кластеров и фактически все геномы обладают семикластерной структурой. Функциональная роль и эволюционные механизмы возникновения таких групп фрагментов требуют специального изучения.

