В 2020 г. было зарегистрировано более 310 тыс. смертей от рака прямой кишки (РПК), что делает данную нозологию лидирующей по показателю летальности среди других онкологических заболеваний [1]. Для облегчения симптомов и локального контроля РПК в настоящее время применяется лучевая терапия (ЛТ) [2]. Несмотря на многолетнее совершенствование алгоритмов ЛТ РПК полный клинический ответ достигается лишь у небольшого числа пациентов, что связано с формированием радиорезистентности злокачественных клеток. За последние 30 лет был проведен внушительный объем исследований по изучению механизмов радиорезистентности и предложен значительный перечень её молекулярных маркеров, ни один из которых, однако, так и не вошёл в клиническую практику. В настоящее время, как никогда ранее, актуально проведение комплексного исследования, интегрирующего различные молекулярные параметры и позволяющего как объяснить ряд молекулярных механизмов радиорезистентности, так и предложить перечень новых маркеров.
В последнее десятилетие сложность человеческого генома была выявлена с помощью высокопроизводительного секвенирования РНК [3]. Было показано существование регуляторной сети конкурирующих эндогенных РНК (ceRNA), состоящей из длинных некодирующих РНК (lncRNA), микроРНК (miRNA) и мРНК.
МикроРНК – это короткие некодирующие РНК, которые регулируют экспрессию генов, катализируя разрушение мРНК либо ингибируя трансляцию мРНК в белок. МикроРНК вносят значительный вклад в инициацию и развитие различных молекулярных событий, включая инициацию онкогенеза, прогрессирование и метастазирование опухолей, что делает микроРНК потенциальными биомаркерами для оценки прогрессирования и прогноза злокачественных опухолей кишечника. Хотя микроРНК регулируют экспрессию генов, кодирующих белки, главным образом посредством деградации или сайленсинга мРНК, появляется все больше свидетельств того, что микроРНК могут взаимодействовать с lncRNA, что, в свою очередь, также обеспечивает регуляцию экспрессии генов-мишеней [4]. Длинные некодирующие РНК (lncRNAs) – это РНК размером более 200 нуклеотидов, регулирующие биологическую активность клеток различными способами, включая транскрипционную регуляцию, посттранскрипционную регуляцию и регуляцию трансляции. lncRNA контролируют посттранскрипционную регуляцию, являясь компонентом сети ceRNAs и выступая в качестве молекулярной «губки» (сорбента) микроРНК [5, 6].
Участие регуляторной сети ceRNA в инициации и прогрессии опухолей кишечника было подтверждено в ряде исследований [7]. Однако роль дисбаланса регуляторной сети ceRNA (lncRNA – miRNA – mRNA) у больных раком прямой кишки и формированием радиорезистентности еще предстоит выяснить [8].
Изучение регуляторной сети микроРНК – длинные некодирующие РНК – мРНК имеет большое значение как для выяснения молекулярных механизмов, лежащих в основе канцерогенеза, так и для создания панели новых биомаркеров [4].
Поэтому целью исследования стал анализ особенностей функционирования регуляторной сети конкурентно-взаимодействующих РНК (lncRNA, miRNA и мРНК) у больных раком прямой кишки, радиорезистентных и чувствительных к лучевой терапии.
Материалы и методы исследования
В исследование было включено 100 пациентов (60 мужчин и 40 женщин в возрасте от 34 до 82 лет, медиана возраста 60,1 ± 5,0 лет) с диагностированным РПК (аденокарциномы), проходивших стационарное лечение в ФГБУ «НМИЦ онкологии» в 2019–2021 гг. ЛТ на линейном ускорителе частиц Novalis TX проводилась по стандартному протоколу (РОД 2.4 Гр, СОД 54.0 Гр). До проведения ЛТ всем пациентам была выполнена видеоколоноскопия (ВКС), во время которой были получены парные фрагменты биопсии условно-нормальных и опухолевых тканей прямой кишки. Эти фрагменты в дальнейшем использовались для выделения РНК.
Выделение суммарных препаратов РНК, анализ уровня мРНК и микроРНК. Фрагменты тканей гомогенизировали в тризоле (QIAzol, Qiagen) при температуре 0–1 °С методом механического воздействия. Из полученных гомогенатов с помощью набора RNeasy Plus Universal Kits (Qiagen) проводили выделение суммарных препаратов РНК. Выделенную РНК обрабатывали ДНКазой-1, тем самым устраняя контаминацию геномной ДНК. На матрице очищенных препаратов РНК осуществляли синтез комплементарной ДНК, используя набор «РЕВЕРТА-Л» (Интерлабсервис, Россия).
Для оценки уровня транскриптов 19 генетических локусов использовали метод ПЦР в реальном времени (Real-Time qPCR, RT-qPCR) (табл. 1). Последовательности высоко-специфичных синтетических олигонуклеотидов были разработаны с использованием программы Primer-BLAST и базы данных GenBank (National Center for Biotechnology Information) (табл. 1).
Таблица 1
Синтетические олигонуклеотиды для определения уровня транскриптов мРНК
|
№ |
Праймеры |
Последовательности праймеров |
№ |
Праймеры |
Последовательности праймеров |
|
1 |
BRCA1(F) |
ACCTGTCTCCACAAAGTGTCT |
19 |
KU70(F) |
ACGTAGAGGGCGTTGATTGG |
|
2 |
BRCA1(R) |
ACACTGTGAAGGCCCTTTTA |
20 |
KU70(R) |
TGGCTACTGCTCACTTTGGCС |
|
3 |
BRCA2(F) |
AGTTGGCTGATGGTGGATGA |
21 |
RAD50(F) |
GCGTGCGGAGTTTTGGAATAG |
|
4 |
BRCA2(R) |
GGATCCACACCTGGAGTGCC |
22 |
RAD50(R) |
TTGAGCAACCTTGGGATCGT |
|
5 |
CASP3(F) |
CTGGAATATCCCTGGACAACAGT |
23 |
RAP80(F) |
GAGTGAGCAGGAAGCTAGGG |
|
6 |
CASP3(R) |
TCGACATCTGTACCAGACCGA |
24 |
RAP80(R) |
AGAAGGCCGGCAAСTATTCA |
|
7 |
CASP8(F) |
CTGAAGCAAACAGCCAGTGC |
27 |
RNF168(F) |
GCCAGTTCGTCTGCTCAGTA |
|
8 |
CASP8(R) |
GACCTCAATTCTGАTCTGCTCAC |
28 |
RNF168(R) |
CTGCCGCCACCTTGCTTAT |
|
9 |
CASP-9(F) |
TGAGACСCTGGACGACATCT |
29 |
H2AX(F) |
AGGCCTCCCAGGAGTACTAA |
|
10 |
CASP-9(R) |
TCCCTTTCACCGAAACAGCA |
30 |
H2AX(R) |
CTGAAGCGGCTCAGCTCTTT |
|
11 |
BAX(F) |
GGGACGAACTGGACAGTAACA |
31 |
ACTB(F) |
AACCGCGAGAAGATGACСC |
|
12 |
BAX(R) |
GCTGCCACTСGGAAAAAGAC |
32 |
ACTB(R) |
AGCACAGCCTGGTAGCAAC |
|
13 |
BCL-2(F) |
GGATCCAGGATAACGGAGGC |
33 |
GAPDH(F) |
GTCAAGGCTGAGAACGGGAA |
|
14 |
BCL-2(R) |
GAAATCAAACAGAGGCCGCA |
34 |
GAPDH(R) |
TCGCCCCACTTGATTTTGGA |
|
15 |
P53(F) |
TTGGAACTCAAGGATGCCCA |
35 |
B2M(F) |
AGATGAGTATGCCTGCCGTG |
|
16 |
P53(R) |
CGGGAGGTAGACTGAСCCT |
36 |
B2M(R) |
CCATGATGCTGCTTACATGTCTC |
|
17 |
MDM2(F) |
TAGGAGATTTGTTTGGCGTGC |
37 |
RBBP-8(F) |
ACCGAGGATTTGGCACTСTG |
|
18 |
MDM2(R) |
CCTGCTGATTGACTACTACСAA |
38 |
RBBP-8(R) |
TCCGAGATTGCCTCGGGATT |
RT-qPCR-смесь состояла из ПЦР-буфера (1х), dNTP (0,2 мМ), MgCl2 (1,5 мМ), синтетических олигонуклеотидов (по 0,6 мМ каждого), Taq-полимеразы (0,1 U/мкл) и комплементарной ДНК (15 нг). Амплификация кДНК проводилась в термоциклере CFX 96 (Bio-Rad) по следующему алгоритму: 94,0 °С – 240 с и далее 39 циклов: 94,0 °С – 9 с, 57 °С – 25 с, 72 °С – 45 с. Относительный уровень транскриптов мРНК (экспрессия генетических локусов) (TA) вычисляли по формуле, учитывающей нормализацию по референсным генам (GAPDH, ACTB и B2M) и уровню мРНК соответствующих генов в образцах нормальной ткани:
TA = E-ΔΔCt = медиана E(C(t)target–C(t)reference)опухолевой ткани / медиана E(C(t)target–C(t)reference)нормальной ткани .[9].
Уровень относительной экспрессии микроРНК и lncRNA также оценивали методом RT-qPCR. Дизайн последовательностей синтетических олигонуклеотидов, необходимых для оценки экспрессии микроРНК, проводили, используя алгоритм Balcells I. Primer-BLAST использовали для дизайна праймеров на длинные некодирующие РНК. В качестве референса для определения экспрессии микроРНК и lncRNA использовали малую некодирующую РНК U6. Для оценки уровня представленности транскриптов микроРНК тотальную РНК конвертировали в кДНК в реакции обратной транскрипции с RT-праймерами одновременно с полиаденилированием [10].
Статистический и биоинформационный анализ выполняли в среде RStudio v. 8.10.173.987 на языке программирования R. Соответствие полученных данных закону нормального распределения оценивали с помощью критерия Шапиро – Уилка. Значимость межгрупповых отличий определяли с использованием критерия Манна – Уитни (поправка Бонферрони). Взаимосвязи исследуемых показателей оценивали с использованием коэффициента корреляции Спирмена (r). Кластерный анализ выполняли с параметрами Hierarchical Clustering/Euclidean distance. Для группировки генетических локусов по биологическим функциям использовали алгоритм FMD (Functional module detection). Q-value вычисляли с помощью одностороннего точного критерия Фишера (поправка Бенджамини – Хохберга) [11].
Для биоинформационного скрининга микроРНК использовали алгоритм машинного обучения random forest (объединяет метод случайных подпространств и метод бэггинга Бреймана), позволяющий прогнозировать сайт связывания мРНК и микроРНК [12]. Участие дифференциально экспрессирующихся микроРНК в ключевых клеточных сигнальных путях оценивали с помощью ORA (Over-Representation Analysis) [13].
Анализ взаимодействия микроРНК и lncRNA проводили следующим образом:
1) из базы Gene Expression Omnibus были извлечены данные HITS-CLIP, PAR-CLIP и CLASH и обработаны с помощью FASTX-Toolkit v0.0.13, и далее проанализированы с использованием PARalyzer v1.1;
2) с помощью UCSC LiftOver Tool все координаты сайтов связывания были преобразованы в сборки hg19, mm9/mm10 и ce6/ce10 соответственно. Геномные координаты консервативных сайтов-мишеней микроРНК, предсказанные TargetScan, miRanda/mirSVR, PITA, Pictar и RNA22, были также собраны и преобразованы в сборки hg19, mm9/mm10 и ce6/ce10 с использованием LiftOver;
3) полученные на шаге 2 координаты сравнивали с ранее описанными кластерами CLIP с использованием BEDTools [4].
Результаты исследования и их обсуждение
Связь уровня мРНК и эффективности ЛТ. Данные по уровню мРНК 19 генов в образцах биопсии опухолевой ткани прямой кишки были подвергнуты кластерному анализу (Hierarchical Clustering/Euclidean distance), позволившему выделить два основных кластера: в кластере-1 у 80 % образцов был повышен уровень мРНК гена CASP-9 и у 75 % снижен уровень мРНК генов BCL-2, BRCA-2, RBBP-8, H2AX; в кластере-2 у 80 % был снижен уровень мРНК CASP-9, а у 82 % повышен уровень H2AX, BCL-2, BRCA-2, RBBP-8 и RAD50.
Комплексный анализ результатов радиотерапии 100 больных РПК дал следующие результаты: у 30 пациентов был получен полный ответ на терапию, у 45 пациентов – частичный ответ, и у 25 пациентов было зафиксировано отсутствие ответа на терапию. В биопсии опухолевого материала у пациентов с полным ответом на терапию был зафиксирован пониженный уровень мРНК генов H2AX и RBBP-8 в 2,50 и 2,00 раза (р = 0,0048), соответственно, и повышенный уровень мРНК CASP-9 в 5,00 раз (р = 0,00078). В опухолевом биопсийном материале у пациентов с частичным ответом/отсутствием ответа на ЛТ был повышен уровень мРНК генов BCL-2, BRCA-2, H2AX, RBBP-8 и RAD50 в 5,20 (р = 0,00008), 2,50 (р = 0,0044), 2,70 (р = 0,0041), 3,20 (р = 0,0011) и 2,10 (р = 0,0051) раза соответственно (рис. 1).
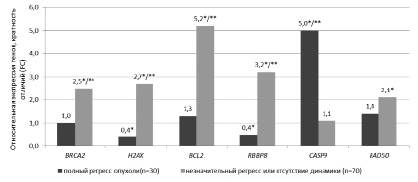
Рис. 1. Относительный уровень мРНК 6 генетических локусов в опухолевой ткани больных РПК с полным ответом на терапию (группа 1, n = 30) и частичным ответом/отсутствием ответа (группа 2, n = 70). * – статистически значимые отличия относительно условно-нормальной ткани прямой кишки (р < 0,05), ** – межгрупповые статистически значимые отличия (р < 0.05)
Таким образом, можно сделать вывод, что уровень мРНК локусов RAD50, BCL-2, BRCA-2, H2AX, CASP-9 и RBBP-8 связан с эффективностью радиотерапии, которая возрастает у больных РПК с гиперэкспрессией гена CASP-9 и гипоэкспрессией генов H2AX и RBBP-8, и, наоборот, эффективность терапии падает при гиперэкспрессии генов RAD50, BCL-2, H2AX, RBBP-8 и BRCA-2.
Связь уровня микроРНК и эффективности ЛТ. Биоинформационный скрининг позволил обнаружить 1927 микроРНК, потенциально взаимодействующих с генетическими локусами H2AX, BCL-2, BRCA-2, RAD50, CASP-9 и RBBP-8. Из 1927 только для 109 микроРНК было подтверждено взаимодействие с соответствующими генами-мишенями в базе данных miRDB (рис. 2). При этом 44 микроРНК образовывали наиболее прочные комплексы с соответствующими генами-мишенями (минимальная свободная энергия взаимодействия в паре микроРНК – мРНК): 18 микроРНК для гена RAD50, 12 микроРНК для CASP-9, 9 микроРНК для гена BCL-2, 2 микроРНК для BRCA-2, 2 микроРНК для H2AX, 1 микроРНК для RBBP-8 (табл. 2).
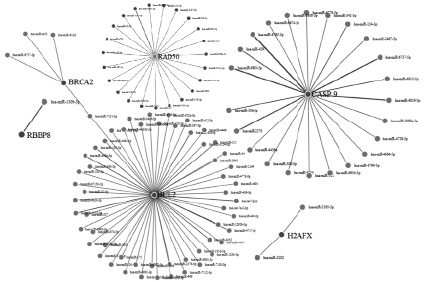
Рис. 2. МикроРНК и их гены-мишени, влияющие на эффективность радиотерапии
Таблица 2
МикроРНК, выявленные при биоинформационном скрининге*
|
МикроРНК |
Ген-мишень |
Координаты в геноме |
Энергия |
miRDB |
|
|
начало |
конец |
||||
|
miRNA-1249 |
BCL-2 |
3468 |
3517 |
-30.1 |
+ |
|
miRNA-6861 |
3474 |
3513 |
-29.6 |
+ |
|
|
miRNA-8052 |
4393 |
4412 |
-29.3 |
+ |
|
|
miRNA-324 |
4680 |
4700 |
-28.6 |
+ |
|
|
miRNA-6820 |
1670 |
1701 |
-28.1 |
+ |
|
|
miRNA-4717 |
2322 |
2364 |
-28.0 |
+ |
|
|
miRNA-3943 |
2750 |
2774 |
-27.8 |
+ |
|
|
miRNA-557 |
2549 |
2584 |
-27.6 |
+ |
|
|
miRNA-4690 |
2147 |
2172 |
-27.3 |
+ |
|
|
miRNA-6757 |
BRCA-2 |
10786 |
10812 |
-25.7 |
+ |
|
miRNA-7151 |
10797 |
10825 |
-25.1 |
+ |
|
|
miRNA-6779 |
CASP-9 |
1482 |
1537 |
-34.3 |
+ |
|
miRNA-1273h |
2555 |
2590 |
-33.1 |
+ |
|
|
miRNA-6812 |
2593 |
2622 |
-30.6 |
+ |
|
|
miRNA-6737 |
1329 |
1347 |
-30.1 |
+ |
|
|
miRNA-661 |
1516 |
1552 |
-29.8 |
+ |
|
|
miRNA-6799 |
2609 |
2656 |
-29.7 |
+ |
|
|
miRNA-6893 |
2442 |
2484 |
-28.5 |
+ |
|
|
miRNA-6819 |
1329 |
1347 |
-27.0 |
+ |
|
|
miRNA-6874 |
2505 |
2535 |
-26.6 |
+ |
|
|
miRNA-4728 |
2906 |
2925 |
-26.3 |
+ |
|
|
miRNA-6808 |
1588 |
1610 |
-26.2 |
+ |
|
|
miRNA-30b |
1967 |
1987 |
-25.6 |
+ |
|
|
miRNA-3202 |
H2AFX |
620 |
639 |
-20.3 |
+ |
|
miRNA-5195 |
1478 |
1504 |
-23.8 |
+ |
|
|
miRNA-130b |
RBBP-8 |
3196 |
3215 |
-23.7 |
+ |
|
miRNA-195 |
RAD50 |
4473 |
4495 |
-21.6 |
+ |
|
miRNA-340 |
7439 |
7465 |
-21.6 |
+ |
|
|
miRNA-497 |
7937 |
7955 |
-21.7 |
+ |
|
|
miRNA-4257 |
4983 |
4995 |
-21.9 |
+ |
|
|
miRNA-5187 |
5263 |
5294 |
-22.1 |
+ |
|
|
miRNA-149 |
6304 |
6325 |
-22.6 |
+ |
|
|
miRNA-6769a |
7407 |
7432 |
-22.7 |
+ |
|
|
miRNA-5010 |
6718 |
6737 |
-23.1 |
+ |
|
|
miRNA-4533 |
4505 |
4523 |
-23.9 |
+ |
|
|
miRNA-6856 |
5076 |
5107 |
-23.9 |
+ |
|
|
miRNA-138-1 |
6188 |
6209 |
-24.2 |
+ |
|
|
miRNA-433 |
5013 |
5034 |
-24.4 |
+ |
|
|
miRNA-3911 |
6964 |
6988 |
-24.4 |
+ |
|
|
miRNA-3691 |
5368 |
5391 |
-24.6 |
+ |
|
|
miRNA-4786 |
6889 |
6915 |
-25.7 |
+ |
|
|
miRNA-6758 |
5054 |
5108 |
-26.0 |
+ |
|
|
miRNA-6865 |
6715 |
6736 |
-29.0 |
+ |
|
|
miRNA-6798 |
5134 |
5170 |
-30.4 |
+ |
|
* – представлены только микроРНК, валидированные в базе данных miRDB с минимальной свободной энергией взаимодействия микроРНК – мРНК.
Для этих 44 микроРНК проведена оценка представленности их транскриптов в тканях у больных РПК. Обнаружена дифференциальная экспрессия ряда микроРНК как межгрупповая, так и относительно нормальной ткани. В группе больных с частичным ответом/отсутствием ответа после радиотерапии (n = 70, низкая эффективность ЛТ) обнаружено снижение уровня miRNA-1249-5p и miRNA-6820-3p в 2,20 (p = 0,0035) и 2,00 (p = 0,0015) раза соответственно относительно нормальной ткани, снижение уровня miRNA-6820-3p в 1,80 (p = 0,0002) раза относительно пациентов с полным ответом на терапию, снижение уровня miRNA-4717-5p, miRNA-3943 и miRNA-557
в 2,50 (p = 0,00085), 3,30 (p = 0,00001) и 2,50 (p = 0,0023) раза соответственно относительно нормальной ткани, снижение уровня miRNA-557 в 2,3 (p = 0,00047) раза относительно пациентов с полным ответом на ЛТ (n = 30). Также выявлено увеличение уровня представленности транскриптов miRNA-4690-5p в 2,1 (p = 0,00017) раза относительно нормальной ткани и снижение miRNA-4717-5p в 1,7 раза (р = 0,000546) относительно нормальной ткани у пациентов с полным ответом на ЛТ (рис. 1).
То есть данные микроРНК, взаимодействующие с геном BCL-2, в опухолевой ткани у больных с плохим ответом на ЛТ, преимущественно гипоэкспрессированы как относительно нормальной ткани, так и относительно опухолевой ткани пациентов с полным ответом на ЛТ. Соответственно, низкий уровень транскриптов данных микроРНК может приводить к увеличению количества транскриптов генетического локуса BCL-2, продукт которого ингибирует каспазы и апоптоз [14]. Это согласуется с полученными данными: низкой эффективностью ЛТ у больных РПК с гиперэкспрессией BCL-2.
В ткани опухоли больных РПК с низкой эффективностью терапии обнаружено увеличение уровня miRNA-1273h в 2,30 раза (p = 0,0044), miRNA-4728 в 4,00 раза (p = 0,00145), miRNA-6819 в 2,90 раза (p = 0,000012), miRNA-6737 в 3,00 раза (p = 0,0004), miRNA-6874в 3,30 раза (p = 0,0051004) и miRNA-6808-5p в 5,01 раз (p < 0,0000047) относительно опухолевой ткани больных с высокой эффективностью терапии. Уровень miRNA-6812 был снижен в опухолевой ткани у больных с полным и частичным ответом на ЛТ в 1,90 (p = 0,0049) и 1,70 (p = 0,0001) раза соответственно относительно нормальной ткани прямой кишки. Геном-мишенью этих микроРНК является CASP-9, соответственно, высокий уровень их транскриптов должен способствовать снижению уровня мРНК инициаторной каспазы каспазы-9, необходимой для запуска апоптоза.
Уровень miRNA-6757 в опухолевой ткани у больных РПК с низкой эффективностью радиотерапии был ниже в 5,00 (p = 0,0009) раз и 4,41 (p = 0,0040) раза относительно собственной нормальной ткани и опухолевой ткани пациентов с высокой эффективностью терапии соответственно. Геном-мишенью данной микроРНК является BRCA-2, и снижение уровня её транскриптов должно приводить к увеличению уровня мРНК BRCA-2 (рис. 1). Уровень miRNA-1273h, miRNA-6737, miRNA-661, miRNA-4728, miRNA-30b и miRNA-6808 был снижен в 2,50 (p = 0,00001), 3,32 (p = 0,0004), 1,71 (p = 0,0005), 5,00 (p = 0,0001), 1,80 (p = 0,0019) и 5,00 (p = 0,00013) раза соответственно в опухолевой ткани пациентов с полным ответом на терапию относительно нормальной ткани.
В группе больных РПК с высокой эффективностью ЛТ выявлены статистически значимые (p < 0,05) изменения в уровне микроРНК miRNA-5195 и miRNA-3202. Так в опухолевой ткани уровень транскриптов miRNA-3202 был в 4,50 раза выше, чем в нормальной ткани, и в 3,00 раза выше, чем в опухолевой ткани у больных РПК с низкой эффективностью терапии. Уровень miRNA-5195 был в 1,80 раза выше по сравнению с нормальной тканью и в 3,60 раза выше, чем уровень в опухолевой ткани у больных РПК с низкой эффективностью ЛТ (рис. 3). Данные микроРНК могут взаимодействовать с мРНК гистонового белка H2AX, запускающего конформационную модификацию хроматина при повреждении ДНК под воздействием ионизирующего излучения [15]. Поэтому высокий уровень miRNA-3202/miRNA-5195 может способствовать снижению уровня мРНК H2AX, и наоборот.
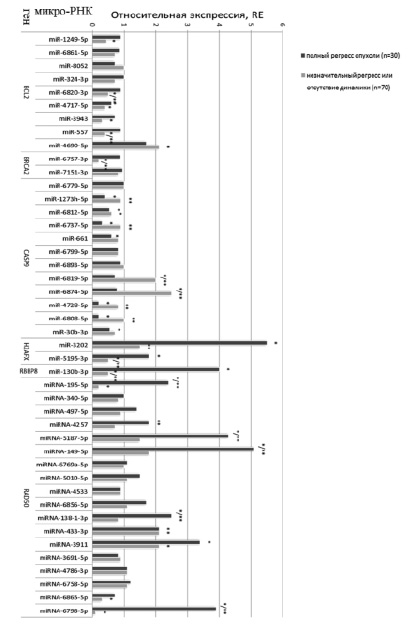
Рис. 3. Транскрипционный профиль микроРНК опухолевой ткани в двух группах больных РПК с разной эффективностью терапии. * – статистически значимые отличия относительно нормальной ткани (р < 0,05), ** – статистически значимые отличия между группами (р < 0,05)
В ходе проведенного исследования также обнаружено, что в опухолевой ткани у больных РПК с высокой эффективностью терапии повышен уровень miRNA-130b в 4,00 раза (p = 0,000014) относительно нормальной ткани и снижен уровень и этой микроРНК в 2,00 раза (p = 0,000195) в опухолевой ткани у больных с низкой эффективностью терапии. Соответственно, у больных первой группы уровень miRNA-130b в 8,00 раза выше (p = 0,00022) по сравнению с уровнем у больных второй группы, что может приводить к снижению уровня мРНК гена RBBP-8 (регулирует пролиферацию клеток) у больных с высокой эффективностью ЛТ и повышению его уровня у больных РПК с низкой эффективностью терапии (рис. 1).
Из 18 микроРНК таргетирующих ген RAD50, в опухолевой ткани прямой кишки аберрантную экспрессию имеют только 8 (miRNA-195-5p, miRNA-5187-5p, miRNA-149-5p, miRNA-138-1-3p, miRNA-433-3p, miRNA-3911, miRNA-6865-5p, miRNA-6798-5p).
Так, экспрессия miRNA-195-5p относительно нормальной ткани прямой кишки в 2,40 (p = 0,0045) раза выше у больных с полным регрессом опухоли и в 5,00 раз (p = 0,0003) ниже у больных с неполным регрессом опухоли. При этом между этими группами отличия в экспрессии miRNA-195-5p составляют 12,00 раз (p = 0,00004) (рис. 3). Экспрессия miRNA-5187-5p и miRNA-149-5p повышена в 4,30 и 5,10 раз (p = 0,00015) в опухолевой ткани у больных с полным ответом на терапию относительно нормальной ткани. Отличие между двумя группами больных по экспрессии этих микроРНК составляет 2,80 раза (p = 0,0001). Экспрессия miRNA-138-1-3p, miRNA-433-3p и miRNA-3911 статистически значимо (p < 0,05) повышена у больных с полным регрессом относительно экспрессии в нормальной ткани в 2,50; 2,10 и 3,40 раза соответственно. Экспрессия miRNA-6798-5p относительно нормальной ткани прямой кишки в 3,90 (p = 0,00075) раза выше у больных с полным регрессом опухоли и в 10,00 раз (p < 0,00022) ниже у больных с частичным регрессом опухоли. При этом между этими группами отличия в экспрессии miRNA-6798-5p составляет 39,00 раз (p = 0,000002) (рис. 3). Экспрессия miRNA-6865-5p снижена в 3,30 раза (p = 0,003) в опухолевой ткани у больных с неполным регрессом опухоли относительно нормальной ткани прямой кишки.
Так как микроРНК имеют большое количество генов-мишеней [16], то для дифференциально экспрессирующихся микроРНК в опухолевых тканях двух групп больных РПК был проведен анализ ORA (Over-Representation Analysis, табл. 3, рис. 4).
Таблица 3
Представленность в сигнальных путях микроРНК с дифференциальной экспрессией
|
Сигнальный путь |
P-value |
МикроРНК |
|
ECM-receptor interaction |
5,53e-6 |
miRNA-195; miRNA-4257; miRNA-149; miRNA-138-1; miRNA-6798; miRNA-6819; miRNA-4728; miRNA-1249; miRNA-557; miRNA-1273h; miRNA-6737; miRNA-6808; miRNA-3202; miRNA-5195; miRNA-130b |
|
Notch signaling pathway |
1,87e-5 |
miRNA-195; miRNA-4257; miRNA-149; miRNA-6798; miRNA-6819; miRNA-4728; miRNA-1249; miRNA-557; miRNA-1273h; miRNA-6737; miRNA-6808; miRNA-3202; miRNA-5195; miRNA-130b |
|
Protein digestion and absorption |
2,98e-4 |
miRNA-195; miRNA-4257; miRNA-6798; miRNA-6819; miRNA-4728; miRNA-1249; miRNA-557; miRNA-1273h; miRNA-6737; miRNA-6808; miRNA-3202; miRNA-5195; miRNA-130b |
|
Glutathione metabolism |
7,10e-4 |
miRNA-195; miRNA-4257; miRNA-5187; miRNA-149; miRNA-138-1; miRNA-6798; miRNA-4728; miRNA-557; miRNA-1273h; miRNA-6808 |
|
Oxidative phosphorylation |
9,45e-4 |
miRNA-195; miRNA-4257; miRNA-149; miRNA-138-1; miRNA-6798; miRNA-6819; miRNA-4728; miRNA-1249; miRNA-1273h; miRNA-6737; miRNA-6808; miRNA-3202; miRNA-5195; miRNA-130b |
|
RIG-I-like receptor signaling pathway |
0,001412 |
miRNA-195; miRNA-4257; miRNA-5187; miRNA-6798; miRNA-6819; miRNA-4728; miRNA-1249; miRNA-557; miRNA-6737; miRNA-6808; miRNA-3202; miRNA-130b |
|
Phosphatidylinositol signaling system |
0,0021463 |
miRNA-195; miRNA-4257; miRNA-5187; miRNA-149; miRNA-6798; miRNA-6819; miRNA-4728; miRNA-1249; miRNA-557; miRNA-1273h; miRNA-6737; miRNA-6808; miRNA-3202; miRNA-130b |
|
Apoptosis – multiple species |
0,0023771 |
miRNA-195; miRNA-4257; miRNA-149; miRNA-6819; miRNA-4728; miRNA-1249; miRNA-557; miRNA-1273h; miRNA-6737; miRNA-6808; miRNA-3202; miRNA-130b |
|
Phagosome |
0,0023569 |
miRNA-195; miRNA-4257; miRNA-149; miRNA-138-1; miRNA-6798; miRNA-6819; miRNA-4728; miRNA-1249; miRNA-557; miRNA-1273h; miRNA-6737; miRNA-6808; miRNA-3202; miRNA-5195; miRNA-130b |
|
Signaling pathways regulating pluripotency of stem cells |
0,0028218 |
miRNA-195; miRNA-4257; miRNA-5187; miRNA-149; miRNA-138-1; miRNA-6798; miRNA-6819; miRNA-4728; miRNA-1249; miRNA-557; miRNA-1273h; miRNA-6737; miRNA-6808; miRNA-3202; miRNA-5195; miRNA-130b |
|
Estrogen signaling pathway |
0,0037057 |
miRNA-195; miRNA-4257; miRNA-5187; miRNA-149; miRNA-6798; miRNA-6819; miRNA-4728; miRNA-1249; miRNA-557; miRNA-1273h; miRNA-6737; miRNA-6808; miRNA-3202; miRNA-5195; miRNA-130b |
|
Inflammatory mediator regulation of TRP channels |
0,0040433 |
miRNA-195; miRNA-5187; miRNA-149; miRNA-6798; miRNA-6819; miRNA-4728; miRNA-1249; miRNA-557; miRNA-1273h; miRNA-6737; miRNA-6808; miRNA-3202; miRNA-130b |
|
Pyrimidine metabolism |
0,0044015 |
miRNA-195; miRNA-4257; miRNA-149; miRNA-6819; miRNA-4728; miRNA-1249; miRNA-1273h; miRNA-6737; miRNA-6808; miRNA-130b |
|
Inositol phosphate metabolism |
0,0046988 |
miRNA-195; miRNA-4257; miRNA-149; miRNA-6819; miRNA-4728; miRNA-1249; miRNA-557; miRNA-1273h; miRNA-6737; miRNA-6808; miRNA-3202; miRNA-130b |
|
Necroptosis |
0,0049473 |
miRNA-195; miRNA-4257; miRNA-5187; miRNA-149; miRNA-6798; miRNA-6819; miRNA-4728; miRNA-1249; miRNA-557; miRNA-1273h; miRNA-6737; miRNA-6808; miRNA-3202; miRNA-5195; miRNA-130b |
|
Cell cycle |
0,0061424 |
miRNA-195; miRNA-4257; miRNA-5187; miRNA-149; miRNA-138-1; miRNA-6798; miRNA-6819; miRNA-4728; miRNA-1249; miRNA-557; miRNA-1273h; miRNA-6737; miRNA-6808; miRNA-3202; miRNA-5195; miRNA-130b |
|
Окончание табл. 3 |
||
|
Сигнальный путь |
P-value |
МикроРНК |
|
ErbB signaling pathway |
0,0059231 |
miRNA-195; miRNA-4257; miRNA-149; miRNA-138-1; miRNA-6798; miRNA-6819; miRNA-4728; miRNA-1249; miRNA-557; miRNA-1273h; miRNA-6737; miRNA-6808; miRNA-3202; miRNA-5195; miRNA-130b |
|
Parathyroid hormone synthesis, secretion and action |
0,0064135 |
miRNA-195; miRNA-4257; miRNA-149; miRNA-138-1; miRNA-6798; miRNA-6819; miRNA-4728; miRNA-1249; miRNA-1273h; miRNA-6737; miRNA-6808; miRNA-3202; miRNA-5195; miRNA-130b |
|
Ribosome biogenesis in eukaryotes |
0,0057282 |
miRNA-195; miRNA-4257; miRNA-149; miRNA-138-1; miRNA-4728; miRNA-1249; miRNA-557; miRNA-1273h; miRNA-6808; miRNA-3202; miRNA-5195; miRNA-130b |
|
VEGF signaling pathway |
0,0077348 |
miRNA-195; miRNA-4257; miRNA-5187; miRNA-149; miRNA-6798; miRNA-6819; miRNA-4728; miRNA-1249; miRNA-6737; miRNA-6808; miRNA-3202; miRNA-130b |
|
C-type lectin receptor signaling pathway |
0,0081841 |
miRNA-195; miRNA-4257; miRNA-5187; miRNA-149; miRNA-6798; miRNA-6819; miRNA-4728; miRNA-1249; miRNA-557; miRNA-1273h; miRNA-6737; miRNA-6808; miRNA-3202; miRNA-130b |
|
Phospholipase D signaling pathway |
0,0088879 |
miRNA-195; miRNA-4257; miRNA-149; miRNA-138-1; miRNA-6798; miRNA-6819; miRNA-4728; miRNA-1249; miRNA-1273h; miRNA-6737; miRNA-6808; miRNA-3202; miRNA-5195; miRNA-130b |
|
Nitrogen metabolism |
0,0097981 |
miRNA-195; miRNA-5187; miRNA-149; miRNA-4728; miRNA-1273h; miRNA-6808 |
Согласно Over-Representation Analysis, дифференциально экспрессирующиеся в двух группах пациентов микроРНК не только оказывают влияние на уровень мРНК генетических локусов BCL-2, CASP-9, BRCA-2, RAD50, H2AX и RBBP-8, но и контролируют ключевые для выживания и пролиферации опухолевых клеток сигнальные пути (табл. 3, рис. 4).
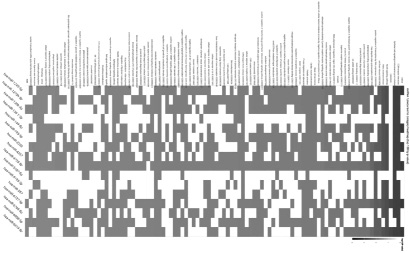
Рис. 4. Визуализация результатов ORA, представленных в табл. 3
Таким образом, радиочувствительность опухолевой ткани прямой кишки, определяющая эффективность лучевой терапии, также связана с уровнем микроРНК miRNA-195, miRNA-4257, miRNA-5187, miRNA-149, miRNA-138, miRNA-6798, miRNA-6820, miRNA-557, miRNA-6757, miRNA-1249, miRNA-1273h, miRNA-6737, miRNA-6819, miRNA-6874, miRNA-4728, miRNA-6808, miRNA-3202, miRNA-5195-3p и miRNA-130b.
Биоинформационный скрининг потенциального взаимодействия микроРНК и длинных некодирующих РНК. Применение биоинформационных алгоритмов позволило выявить для пяти дифференциально экспрессирующихся микроРНК (miR-130b, miR-149, miR-195, miR-1249, miR-5195) взаимодействие с 202 длинными некодирующими РНК (табл. 4).
Таблица 4
Перечень длинных некодирующих РНК и взаимодействующих с ними микроРНК
|
МикроРНК |
Lncrna-мишень |
Последовательность микроРНК |
Выравнивание |
Последовательность длинной некодирующей РНК |
|
hsa-miR-130b-3p |
LINC01087 |
uacGGGAAAGUAGUAACGUGAc |
:| | | : ||||||||| |
uauUCGUCUGGACAUUGCACUu |
|
AC016717.2 |
uaCGGGAAAGUAGUAACGUGAc |
|:|:| |:||| ||||||| |
agGUCUUGUUAUC-UUGCACUa |
|
|
AC109588.1 |
uacgggaaagUAGUAACGUGAc |
| | ||||||| |
uacuuaauaaAACCUUGCACUc |
|
|
AC124016.2 |
uacgggAAAGUAGUAACGUGAc |
|||:| : ||||||| |
auagggUUUUAAU-UUGCACUg |
|
|
LINC01091 |
uacgggaaAGUAGUAACGUGAc |
| :|| ||||||| |
gccuaggcUGGUC-UUGCACUc |
|
|
AL591030.1 |
uacgggaAAGUAGUAACGUGAc |
| :||: ||||||| |
gacagaaUAUAUU-UUGCACUa |
|
|
AP003469.4 |
uacgggaaaguagUAACGUGAc |
|||||||| |
uauucagccuggaAUUGCACUa |
|
|
AL845472.1 |
uaCGGGA--AAGUAGUAACGUGAc |
|||:| | | | :||||||| |
ugGCCUUCAUCCCUAGUUGCACUa |
|
|
PCAT5 |
uacGGGAAAGUAGU--AACGUGAc |
|||| ::||| ||||||| |
ucuCCCUGCUGUCACUUUGCACUu |
|
|
LINC00839 |
uacGGGA--AA- GUAGUAACGUGAc |
|||| || | | ||||||| |
uccCCCUGAUUCCCACUUUGCACUu |
|
|
NEAT1 |
uacGGGAAAGUAGUAACGUGAc |
|: || ::|| ||||||| |
ggaCUGUUCUGUCCUUGCACUg |
|
|
LINC01089 |
uacGGGAAAGUAGUAACGUGAc |
:|| | ||||||||| |
gcuUCCAACCUCCAUUGCACUg |
|
|
AL355338.1 |
uacgggaAAGUAGUAACGUGAc |
| || ||||||||| |
cugagacUGCACCAUUGCACUc |
|
|
VASH1-AS1 |
uacgggaaAGUA--GUAACGUGAc |
|||| ||||||||| |
agccgagaUCAUGCCAUUGCACUc |
|
|
AC142472.1 |
uaCGGGAAAGUAGU--- AACGUGAc |
| | || || || ||||||| |
cuGACAUUCCAGCACGUUUGCACUg |
|
|
LINC01476 |
uacGGGAAAGUAGU-AACGUGAc |
|| | ||| | ||||||| |
aucCCAUGACAUGAUUUGCACUc |
|
|
LINC00667 |
uacGGGAAAGUAGUAACGUGAc |
|:|| | : | ||||||| |
aauCUCUAU-GCCUUUGCACUg |
|
|
ILF3-AS1 |
uacgggAAAGUAGUAACGUGAc |
|| | ||||||||| |
ccgagaUUGCUCCAUUGCACUc |
|
|
CTD-3093M3.1 |
uacgggaaaguaguAACGUGAc |
||||||| |
uauuuguaaaaaauUUGCACUu |
|
|
LINC02575 |
uacgggaaaGUAGUAACGUGAc |
|| | ||||||| |
acucaauaaCA-CUUUGCACUc |
|
|
XIST |
uacgggaAAGUAGU-AACGUGAc |
||| ||| ||||||| |
uaaacaaUUC-UCAGUUGCACUu |
|
|
hsa-miR-149-5p |
AL645608.1 |
ccCUCACUUCUGUGC- CUCGGUCu |
|: ||:||:|: | ||||||| |
cgGGAUGGAGGCGGGAGAGCCAGg |
|
LINC01356 |
cccucacuucUGUGCCUCGGUCu |
|| |||||||| |
uuuaauacccACCAGGAGCCAGu |
|
|
BLACAT1 |
cccUCACUUCUGUGCCUCGGUCu |
| ||| :| |||||||| |
agcACUGA--GCUGGGAGCCAGg |
|
|
AC007878.1 |
cccucACUUCUGUGCCUCGGUCu |
||:: | || ||||||| |
cagccUGGGCA-ACAGAGCCAGg |
|
|
AC009237.14 |
cccucacUUCUGUGCCUCGGUCu |
|||:| ||||||| |
gaagcacAAGGCUGAGAGCCAGu |
|
|
MIR4435-2HG |
cccUCACUUCUG--UG- CCUCGGUCu |
||| | :| |: |||||||| |
uccAGUAAUUGCUAAUGGGAGCCAGu |
|
|
AC079922.2 |
cccUCAC-UUCUGUGCCUCGGUCu |
|||| : || : ||||||| |
ugcAGUGUGUCACCUUGAGCCAGu |
|
|
AC097468.3 |
ccCUCACUUCUGUGCCUCGGUCu |
|| ||| | |||||||| |
auGAAUGACCCAAAGGAGCCAGg |
|
|
AC109779.1 |
ccCUCACUUCUGUGC---CUCGGUCu |
|: || |::|:| ||||||| |
guGGCUGCUGGUAUGCAUGAGCCAGc |
|
|
AC131235.4 |
cccucACUUCUGUGC---CUCGGUCu |
|| |::|:| ||||||| |
uuauaUGCUGGUAUGCCAGAGCCAGa |
|
|
MUC20-OT1 |
cccucACUUCUGUGCCUCGGUCu |
| ::|::| |||||||| |
uguuuUAGGGGUA-GGAGCCAGg |
|
|
AC024132.1 |
cccUC-ACUUCUGUGCCUCGGUCu |
|| ||:| : ||||||| |
cucAGCUGGACCAGAAGAGCCAGa |
|
|
SNHG8 |
cccuCACUU--CUGU--GCCUCGGUCu |
| ||| | || : ||||||| |
ccccGAGAACCGUCAGUUUGAGCCAGa |
|
|
SCAMP1-AS1 |
cccUC-ACUUC--UGUGCCUCGGUCu |
|| ||||| ||| |||||||| |
cuuAGAUGAAGCUACAGGGAGCCAGa |
|
|
LINC01554 |
cccucacuucuguGCCUCGGUCu |
| ||||||| |
cgcagcuucacucCUGAGCCAGc |
|
|
LINC01848 |
ccCUCACUUCUGUGCCUCGGUCu |
||| |:|| |:|||||||| |
uuGAG-GGAGCAAUGGAGCCAGc |
|
|
AC094104.1 |
cccuCACUUCUGUGCCUCGGUCu |
|||::|| | ||||||| |
gaacGUGGGGA-AGUGAGCCAGg |
|
|
C5orf66-AS1 |
cccuCACUUCUGUGCCUCGGUCu |
||| || | ||||||| |
aaccGUGCAGUGCCAGAGCCAGa |
|
|
AL390955.2 |
cccucACUUCUGUGCCUCGGUCu |
||:||: | ||||||| |
cucccUGGAGG-A--GAGCCAGc |
|
|
AC092171.3 |
ccCUCACUUCUGUGCCUCGGUCu |
||| |:||:| ||||||||| |
ggGAGCGGAGGC-CGGAGCCAGc |
|
|
AC005537.1 |
ccCUCA-CUUCUGUGCCUCGGUCu |
| || |:| | | ||||||| |
ggGCGUAGGACCCUCCGAGCCAGg |
|
|
AC078846.1 |
cccucacUUCUGUG--CCUCGGUCu |
|||:::| ||||||| |
accaaaaAAGGUGCCUUGAGCCAGa |
|
|
Продолжение табл. 4 |
||||
|
МикроРНК |
Lncrna-мишень |
Последовательность микроРНК |
Выравнивание |
Последовательность длинной некодирующей РНК |
|
AL158152.1 |
cccUCACU-UCUG--UGCCUCGGUCu |
| ||| ||:: |: ||||||| |
uucAUUGAUAGGUUGAUUGAGCCAGa |
|
|
AL365199.1 |
cccucaCUUCUGU-GCCUCGGUCu |
|: |::| :|||||||| |
acccaaGGUGGUAUUGGAGCCAGg |
|
|
LINC01468 |
cccucaCUUCUGUGC---CUCGGUCu |
|||||:: | ||||||| |
aaacugGAAGAUGAGGAAGAGCCAGc |
|
|
OLMALINC |
cccucacuucUGUGCCUCGGUCu |
| :| ||||||| |
gcuccucgccAGGCUGAGCCAGg |
|
|
C10orf91 |
cccucacuucugugcCUCGGUCu |
||||||| |
cacaggcggcuggaaGAGCCAGu |
|
|
NEAT1 |
cccUCACUUC-UGUG-C-CUCGGUCu |
|| ||| || | | ||||||| |
aaaAGGAAAGUACCCUGAGAGCCAGu |
|
|
MALAT1 |
ccCUCACUUCUGUGCCUCGGUCu |
|| :|| | |||||||| |
agGAAAAGAGUCCAGGAGCCAGu |
|
|
AC012531.1 |
cccucacuucuguGCCUCGGUCu |
| ||||||| |
uccucgucgcuauCUGAGCCAGg |
|
|
AC023794.6 |
cccucacuucuGUGCCUCGGUCu |
||| ||||||| |
ccaccccugucCACAGAGCCAGg |
|
|
HELLPAR |
cccUCACUU-CUGUGCCUCGGUCu |
||| || ||:: ||||||| |
aaaAGUUAAUGAUGGUGAGCCAGg |
|
|
AC125603.2 |
cccucacuucuguGCCUCGGUCu |
| ||||||| |
ugcagcuucacucCUGAGCCAGc |
|
|
LINC00943 |
ccCUCACUUCUGUGCCUCGGUCu |
|: ||| :|| |||||||| |
caGGCUGA--GCAAGGAGCCAGu |
|
|
LINC01257 |
cccUCACUUCUGUGCCUCGGUCu |
:||| :|: :| ||||||| |
cgcGGUGCGGGAGCAGAGCCAGc |
|
|
AF111169.1 |
cccucaCUUCUG-UGCCUCGGUCu |
||| || : |||||||| |
ggggcaGAACACUGGGGAGCCAGg |
|
|
VASH1-AS1 |
ccCU-CACUUCUGU-GCCUCGGUCu |
|| || : :|| | ||||||| |
caGAUGUUGCUGCAGCUGAGCCAGa |
|
|
LINC02288 |
cccuCACUUCUGUGCCUCGGUCu |
|| ||| | |||||||| |
cccuGUCUAGA-A-GGAGCCAGu |
|
|
DRAIC |
ccCUCAC-UUCUGUGCCUCGGUCu |
||| | : |:||| ||||||| |
cuGAGAGCGCGGCAC-GAGCCAGa |
|
|
LINC02188 |
cccUCACUUCUGUGCCUCGGUCu |
:||| ||| : ||||||| |
gaaGGUGUAGA-GGAGAGCCAGg |
|
|
RP1-178F10.3 |
cccucacuucuGUGCCUCGGUCu |
||:|||||||| |
aucgaggucauCAUGGAGCCAGc |
|
|
AC068512.1 |
cccucacuUCUGUGCCUCGGUCu |
|| : :|||||||| |
ggcccuacAGCUUUGGAGCCAGg |
|
|
AC006504.5 |
cccUCACUUC-UGUGCCUCGGUCu |
|| | | :: | ||||||| |
uccAGGCAUGUGUCCUGAGCCAGg |
|
|
AL121894.2 |
cccucACUUCUGUGCCUCGGUCu |
|||:| || ||||||| |
ccccaUGAGGCCAGUGAGCCAGa |
|
|
LINC00895 |
cccucaCUUCUG-UGCCUCGGUCu |
||:|:| : ||||||| |
uuguuaGAGGGCUGAAGAGCCAGc |
|
|
OGFRP1 |
ccCUCACUUCUGUGC-CUCGGUCu |
|| | : | | | ||||||| |
guGACUUGUGCGAAGUGAGCCAGg |
|
|
MIRLET7BHG |
cccUCACUUC-UGUGCCUCGGUCu |
| |||:| || ||||||| |
gaaACUGAGGCCCAGAGAGCCAGg |
|
|
XIST |
cccucacuUCUGUGCCUCGGUCu |
|| | : ||||||| |
ccugggccAGUC-UUGAGCCAGc |
|
|
FTX |
cccucaCUUCUGUGCCUCGGUCu |
||| : | ||||||| |
ugcgcaGAACUUCCCGAGCCAGc |
|
|
LINC00630 |
cccucacuucugugcCUCGGUCu |
||||||| |
ugucugucaccuuaaGAGCCAGu |
|
|
DANT2 |
cccucacuucuguGCCUCGGUCu |
| ||||||| |
uauaaacgcucucCUGAGCCAGu |
|
|
MIR503HG |
cccucacuucuGUGCCUCGGUCu |
| :|||||||| |
guacgguucccCCUGGAGCCAGa |
|
|
hsa-miR-195-5p |
AL645608.3 |
cgGUUAUAAAGAC---ACGACGAu |
| | | |||| ||||||| |
ccCCAGAGCUCUGCCCUGCUGCUg |
|
AL031281.2 |
cggUUAUA-AAGACACGACGAu |
| ||| | || ||||||| |
gugAUUAUCUCCUCUGCUGCUa |
|
|
AL691459.1 |
cgguuauaaagacACGACGAu |
||||||| |
uuaggaaagagaaUGCUGCUa |
|
|
RP6-206I17.2 |
cgGUUAUAAAGACACGACGAu |
|||| || | ||||||| |
ccCAAUUUUAC--UGCUGCUg |
|
|
RASAL2-AS1 |
cgguuauaaAGACACGACGAu |
||| ||||||| |
cgguccgggUCUCUGCUGCUc |
|
|
MIR29B2CHG |
cgguuauaaagacACGACGAu |
||||||| |
acugcagaggaacUGCUGCUc |
|
|
LINC01703 |
cgguuauaaagacACGACGAu |
||||||| |
cgugauuagagauUGCUGCUg |
|
|
AC108488.1 |
cgguuauaaagacACGACGAu |
||||||| |
gcuuugugaagaaUGCUGCUg |
|
|
AC010969.2 |
cggUUAUAAAGACACGACGAu |
::| || || ||||||| |
uggGGUCUUGCUAUGCUGCUc |
|
|
AC104794.2 |
cgguuauaaaGACACGACGAu |
|| ||||||| |
gcuaugcacgCU-UGCUGCUa |
|
|
AC073046.1 |
cgguuauAAAGAC-----ACGACGAu |
|||||| ||||||| |
gaucccuUUUCUGAGGGCUGCUGCUg |
|
|
AC019069.1 |
cgGUUAUAAAGACACGACGAu |
| :|| || ||||||| |
gcCUGUA-GUCCCUGCUGCUc |
|
|
TEX41 |
cgguuauaaagacACGACGAu |
||||||| |
agaugcauaaaauUGCUGCUu |
|
|
LINC02478 |
cgguuauAAAGACACGACGAu |
||||| ||||||| |
uuccuccUUUCU-UGCUGCUa |
|
|
AC016717.2 |
cgGUUAUAAAGACACGACGAu |
||||| ||| |||||||| |
agCAAUAGUUCAGUGCUGCUc |
|
|
AC097634.1 |
cggUUAUA-AAGAC--ACGACGAu |
|| || ||: | ||||||| |
aaaAAGAUCUUUAGAUUGCUGCUa |
|
|
LINC02035 |
cgguuauaaagacACGACGAu |
||||||| |
gcuuuacuuaaaaUGCUGCUu |
|
|
AC107068.1 |
cgguUAUAAAGAC---ACGACGAu |
|| | ||| ||||||| |
uguuAUCAUCCUGGGAUGCUGCUu |
|
|
LINC02434 |
cgguuauaaagacACGACGAu |
||||||| |
ggagggaccagauUGCUGCUu |
|
|
AC106799.3 |
cgGUU-AUAAAGACACGACGAu |
||| | || || ||||||| |
aaCAAUUCUUCCUUUGCUGCUu |
|
|
AC093297.2 |
cgGUUAUAAAGAC---ACGACGAu |
||| |:|| ||||||| |
ugCAACCGCUUUGGAUUGCUGCUu |
|
|
AC010245.2 |
cgguuaUAAAGACACGACGAu |
||| | ||||||| |
cuuuccAUUGAUCUGCUGCUg |
|
|
EPB41L4A-AS1 |
cgguuauaaagacACGACGAu |
||||||| |
cugaaguguagacUGCUGCUa |
|
|
Продолжение табл. 4 |
||||
|
МикроРНК |
Lncrna-мишень |
Последовательность микроРНК |
Выравнивание |
Последовательность длинной некодирующей РНК |
|
LINC01184 |
cgGUUAUA--AAGAC-ACGACGAu |
:|:|:| || || ||||||| |
uuUAGUGUCAUUAUGUUGCUGCUa |
|
|
HCG17 |
cgguuauaaagacACGACGAu |
||||||| |
ugcuuugcccgccUGCUGCUg |
|
|
LINC00473 |
cgguuaUAAA-GAC--ACGACGAu |
|||| ||| ||||||| |
agggccAUUUCCUGUUUGCUGCUu |
|
|
AC004852.2 |
cgGUUAUAA-AGACACGACGAu |
||| ||| | ||||||| |
uuCAAAAUUGCCACUGCUGCUg |
|
|
AC005537.1 |
cgGUUAUAAAGACACGACGAu |
||||| : ||||||| |
cuCAAUAAAAU--UGCUGCUa |
|
|
AC118758.3 |
cgguuauaaagACACGACGAu |
| ||||||| |
caggccuagcgUUUGCUGCUu |
|
|
AC003092.1 |
cgGUUA-UA-AAGACACGACGAu |
|| | || | || ||||||| |
cuCACUCAUCUGCUAUGCUGCUg |
|
|
CASC9 |
cgguuauaaagACACGACGAu |
| ||||||| |
agauggacacaUUUGCUGCUu |
|
|
RNF139-AS1 |
cgguuAUAAAGACACGACGAu |
||||| ||||||| |
uugguUAUUU-AAUGCUGCUu |
|
|
PVT1 |
cgguuauaAAGAC-ACGACGAu |
| ||| ||||||| |
uuugcuucUCCUGUUGCUGCUa |
|
|
C9orf163 |
cgguuauaaaGACACGACGAu |
|| ||||||| |
ccugguggggCUUUGCUGCUu |
|
|
LINC00707 |
cgguUAUAAAGAC---ACGACGAu |
| | |||| ||||||| |
cugcAGACAUCUGGACUGCUGCUu |
|
|
LINC00843 |
cgguuauAAAGACACGACGAu |
| | ||||||||| |
uccccguUCUGUGUGCUGCUg |
|
|
AC022400.5 |
cgguuauAAAGACACGACGAu |
| | ||||||||| |
uccccguUCUGUGUGCUGCUg |
|
|
AL132656.2 |
cgGUUAUAAAGACACGACGAu |
| | ||| | ||||||| |
aaCCACAUUCC--UGCUGCUg |
|
|
AL160290.2 |
cggUUAUAAAGACACGACGAu |
|: | || |||||||| |
cagAGGAGCUCCGUGCUGCUa |
|
|
AL158835.1 |
cgguUAUAAAGACACGACGAu |
||||| | ||||||| |
auucAUAUUCAUUUGCUGCUg |
|
|
NEAT1 |
cgguuauaaagacACGACGAu |
||||||| |
cucgccuucacgcUGCUGCUg |
|
|
AC008124.1 |
cggUUA--UAAAGACACGACGAu |
||| | | || ||||||| |
caaAAUCCAAUCCU-UGCUGCUg |
|
|
AC024884.2 |
cgguuauaAAGACACGACGAu |
| || ||||||| |
cagcuuugUACUAUGCUGCUc |
|
|
HELLPAR |
cgguuauaaAGACACGACGAu |
|| ||||||| |
ugcuucaaaUCAUUGCUGCUu |
|
|
AC055713.1 |
cgguuAUAA-AGACACGACGAu |
|||| |: ||||||| |
uuggcUAUUAUUAAUGCUGCUa |
|
|
LINC00943 |
cgguuauAAAGACACGACGAu |
|| || ||||||| |
uagucacUUCCUCUGCUGCUg |
|
|
AL139327.2 |
cgguuAUAAAGAC-ACGACGAu |
| || | | ||||||| |
uuuuuUUUUCCAGAUGCUGCUg |
|
|
AL138820.1 |
cgguuauAAAGACACGACGAu |
|| || ||||||| |
ucuugccUUACU-UGCUGCUu |
|
|
LINC00639 |
cgGUUAUA-AAGACACGACGAu |
:|| || | | ||||||| |
ugUAAGAUGUGAUUUGCUGCUc |
|
|
AL355916.1 |
cgguuauaaAGACACGACGAu |
|| |||||||| |
ucucugaccUCAGUGCUGCUg |
|
|
AL049775.1 |
cgguuauaaagacACGACGAu |
||||||| |
gaggcugugggaaUGCUGCUu |
|
|
MEG3 |
cgGU-UAUAAAGACACGACGAu |
|| ||:| | ||||||| |
caCACAUGUGGCCUUGCUGCUg |
|
|
MEG8 |
cgguuauaaaGACACGACGAu |
|| ||||||| |
auuuaaccacCU-UGCUGCUg |
|
|
AL117190.1 |
cgguuauaaaGACACGACGAu |
|| ||||||| |
auuuaaccacCU-UGCUGCUg |
|
|
LINC00638 |
cgGUUAUAAAGACACGACGAu |
| | | || ||||||| |
gcCCAGCUGUCCAUGCUGCUg |
|
|
FAM30A |
cgguuauaaagacACGACGAu |
||||||| |
agagggaaagggaUGCUGCUg |
|
|
AC012236.1 |
cgguuauaaagacACGACGAu |
||||||| |
agagaggggcaccUGCUGCUa |
|
|
AC020661.1 |
cggUUAUAAAGACACGACGAu |
|| | | ||||||||| |
ugaAAGACAU-UGUGCUGCUg |
|
|
IQCH-AS1 |
cgGUUAUAAAGA------CACGACGAu |
||||: || | |||||||| |
ucCAAUGAUUGUGGUGACGUGCUGCUa |
|
|
AC068338.2 |
cggUUAUAAAGACACGACGAu |
|| || | ||||||| |
acaAAGAUAAC-CUGCUGCUg |
|
|
LINC02128 |
cgguuauaaaGAC--ACGACGAu |
||| ||||||| |
uccgucagacCUGCCUGCUGCUu |
|
|
LINC00922 |
cgguuauaaagacACGACGAu |
||||||| |
agcccugugagguUGCUGCUg |
|
|
AC040162.3 |
cgGUUAUAAAGACACGACGAu |
| ||| | ||||||| |
ugCUAUA-AACACUGCUGCUu |
|
|
LINC01572 |
cgguUAUAAAGAC-ACGACGAu |
|||| | | ||||||| |
uaucAUAUACCCGCUGCUGCUa |
|
|
AC133540.1 |
cgguuauaAAGAC--ACGACGAu |
| ||| ||||||| |
ccuuggccUGCUGGCUGCUGCUc |
|
|
FENDRR |
cggUUAUAAAGAC-----ACGACGAu |
|||| | ||| ||||||| |
aggAAUA-UCCUGCAACCUGCUGCUc |
|
|
AC126696.3 |
cgguuauaaAGAC-ACGACGAu |
|| | ||||||| |
aaagcuccaUCGGCUGCUGCUg |
|
|
LINC00324 |
cgguuauaAAGACACGACGAu |
|| | ||||||| |
uccgcccgUUAU-UGCUGCUc |
|
|
RP1-178F10.3 |
cgguuauaaagacACGACGAu |
||||||| |
uucuguuggcgccUGCUGCUg |
|
|
AC129926.1 |
cgGUU-AUAAAGAC-ACGACGAu |
||: |: |||| ||||||| |
ucCAGCUGCCUCUGCUGCUGCUg |
|
|
SNHG25 |
cgguUAUAAAGACACGACGAu |
|| | || ||||||| |
ugucAUCGUCCU-UGCUGCUu |
|
|
LINC00511 |
cggUUAUAAA-GACACGACGAu |
|:|:||| || ||||||| |
gaaAGUGUUUCCUCUGCUGCUa |
|
|
AC090772.3 |
cggUUAUAAAGAC--ACGACGAu |
| | || || ||||||| |
gugACUUUUGGUGGAUGCUGCUg |
|
|
AC068473.5 |
cgguuauAAAGACACGACGAu |
| ||| ||||||| |
uuuugacUAUCUCUGCUGCUu |
|
|
LINC00662 |
cggUUAUAAAGAC-ACGACGAu |
|||: | ||| ||||||| |
cugAAUGGUACUGCUGCUGCUg |
|
|
SCGB1B2P |
cgguuauaaagacACGACGAu |
||||||| |
cuagcucccgcccUGCUGCUg |
|
|
LINC00665 |
cgGUUAUAAAG-ACACGACGAu |
:| |: | | | ||||||| |
gaUACUGCUCCUUUUGCUGCUu |
|
|
AC010624.2 |
cgguuauaaaGAC-ACGACGAu |
||| ||||||| |
gguccccccaCUGCUGCUGCUa |
|
|
AC005261.1 |
cgguuauaaagacACGACGAu |
||||||| |
cacugauuaaaacUGCUGCUu |
|
|
AL035252.3 |
cgguuauaaAGACACGACGAu |
|| ||||||| |
cucuaggccUCACUGCUGCUu |
|
|
Окончание табл. 4 |
||||
|
МикроРНК |
Lncrna-мишень |
Последовательность микроРНК |
Выравнивание |
Последовательность длинной некодирующей РНК |
|
SMIM25 |
cgguuauaaagacACGACGAu |
||||||| |
ccacugccaccacUGCUGCUc |
|
|
AP001432.1 |
cgGUUAUAAAGACACGACGAu |
||:| || ||||||| |
cuCAGU-CAUCACUGCUGCUu |
|
|
LINC01547 |
cgguuauaAAGACACGACGAu |
| || ||||||| |
cggccccgUCCUCUGCUGCUg |
|
|
AP001476.1 |
cgguuauaaagacACGACGAu |
||||||| |
cgggcgagcccgaUGCUGCUc |
|
|
AP000547.3 |
cgGUUAUAAAGAC-ACGACGAu |
|::|| ||| ||||||| |
ccCGGUA--CCUGCUGCUGCUc |
|
|
BX890604.1 |
cgguuauaaagacACGACGAu |
||||||| |
cgaggccggcggcUGCUGCUa |
|
|
FAM239A |
cgguuauaaagacACGACGAu |
||||||| |
cgaggccggcggcUGCUGCUa |
|
|
FAM239B |
cgguuauaaagacACGACGAu |
||||||| |
uaccuggggccguUGCUGCUu |
|
|
XIST |
cgGUUAUAAAGACACGACGAu |
|||| | | | ||||||| |
gcCAAUUUGUGUUUGCUGCUc |
|
|
MIR503HG |
cggUUAUAAAGACACGACGAu |
|||: | | ||||||| |
uugAAUGACUGUAUGCUGCUu |
|
|
hsa-miR-1249-3p |
AC239800.3 |
acuucuuccccccCUUCCCGCa |
|||||||| |
gacucugaaaccaGAAGGGCGg |
|
AL391069.4 |
acuucuuccccccCUUCCCGCa |
|||||||| |
cgcuggguuccuaGAAGGGCGc |
|
|
TERC |
acuucuuccccCCCUUCCCGCa |
| |||||||| |
uaacccuaacuGAGAAGGGCGu |
|
|
AC008392.1 |
acuucuucccccccUUCCCGCa |
||||||| |
ccucaguacuguaaAAGGGCGg |
|
|
LINC01547 |
acuucuucccccccUUCCCGCa |
||||||| |
ucggcuucagaauaAAGGGCGc |
|
|
XIST |
acuucuucccccccUUCCCGCa |
||||||| |
ucugcaguuaagcuAAGGGCGu |
|
|
hsa-miR-5195-3p |
LINC01355 |
ucGGGGGAGUCU-C-UUGACCUa |
| |::| | | | ||||||| |
caCACUUUAAAAUGUAACUGGAu |
|
AL365181.2 |
ucgggggagUCU-CUUGACCUa |
:|| |||||||| |
agaacagcgGGAGGAACUGGAg |
|
|
AC097634.1 |
ucGGGGGAGUC--U-CUUGACCUa |
|::: |||| | ||||||| |
uuCUUUAUCAGUAAUCAACUGGAa |
|
|
MUC20-OT1 |
ucggggGAGUCUCUUGACCUa |
|| :| ||||||| |
caugggCUGGGCCAACUGGAa |
|
|
AC093297.2 |
ucgggggAGUCUC--UUGACCUa |
| :|:| ||||||| |
uuuagaaUAGGGGCAAACUGGAa |
|
|
AL008729.2 |
ucGGGGGAGUCUC-UUGACCUa |
| ||| : || ||||||| |
gaCACCCAGGAAGCAACUGGAa |
|
|
AC008080.1 |
ucgGGGGAGUCUCUUGACCUa |
|:||| || ||||||| |
cagCUCCU--GA-AACUGGAa |
|
|
AC016831.5 |
ucgggGGAGUCUCUUGACCUa |
|:|| | ||||||| |
cuuugCUUCCCAUAACUGGAa |
|
|
CASC9 |
ucgGGGGAGUCUCUUGACCUa |
||::|| ||||||| |
gaaCCUUUC----AACUGGAu |
|
|
PVT1 |
ucggGGGAGUCUCUUGACCUa |
|| || |:|||||||| |
guugCCAUCUGGGAACUGGAu |
|
|
C9orf170 |
ucgggggagUCUCUUGACCUa |
||||||||||| |
augugggggAGAGAACUGGAg |
|
|
LINC00707 |
ucgggggaGUCUCUUGACCUa |
|||| ||||||| |
ugcagugcCAGAAAACUGGAa |
|
|
LINC01468 |
ucGGGGGAGUCUCUUGACCUa |
|:|| |:: | ||||||| |
caCUCCAUUGUA-AACUGGAa |
|
|
MALAT1 |
ucgggggaGUCUCUUGACCUa |
::|| ||||||| |
guuugaagUGGAAAACUGGAa |
|
|
AP003486.1 |
ucgggggagucuCUUGACCUa |
|||||||| |
uacaaagaaguuGAACUGGAu |
|
|
AC008124.1 |
ucgggggAGUCUCUUGACCUa |
| ||| ||||||| |
gaugugaUGAGA-AACUGGAg |
|
|
AC083805.1 |
ucGGGGGAGUCUCUUGACCUa |
|| ||: ||||||| |
gcCCAACUUUCCUAACUGGAg |
|
|
ATP2B1-AS1 |
ucGGGGGAGUCUCUUGACCUa |
|:: |: ||||||||| |
cuCUUAAUUCCAGAACUGGAa |
|
|
HELLPAR |
ucgggggagucucUUGACCUa |
||||||| |
cauacguaucauuAACUGGAa |
|
|
MAPKAPK5-AS1 |
ucGGGGGAGUCUCUUGACCUa |
|:||:|| | ||||||| |
uaCUCCUUCUGCCAACUGGAa |
|
|
LINC01089 |
ucgggggagucUCUUGACCUa |
| ||||||| |
gaaacuagauuAAAACUGGAa |
|
|
MEG3 |
ucGGGGGAGUCUCUUGACCUa |
||: || || ||||||| |
caCCUGCU-AGCAAACUGGAg |
|
|
IQCH-AS1 |
ucgggggagucucUUGACCUa |
||||||| |
uagaaaauacaccAACUGGAa |
|
|
LINC00052 |
ucgggggagucucUUGACCUa |
||||||| |
aaaaccaaaugccAACUGGAg |
|
|
AC092127.1 |
ucgGGGGAGUCU--C---UUGACCUa |
||::|::|| | ||||||| |
agaCCUUUUGGAAGGCUUAACUGGAu |
|
|
AC005920.3 |
ucgggggaGUCUCUUGACCUa |
::| |||||||| |
agagugaaUGGUGAACUGGAc |
|
|
LINC00662 |
ucgggggagucUCUUGACCUa |
| ||||||| |
auuuuauaauaAAAACUGGAa |
|
|
AC012313.1 |
ucgggggagucucUUGACCUa |
||||||| |
caugaguucccccAACUGGAg |
|
|
BX640515.1 |
ucgGGGGAGUCU---CUUGACCUa |
|::|| ||: ||||||| |
uugCUUCUAAGGAUUAAACUGGAa |
|
|
LINC01671 |
ucggGGGAGUCUCUUGACCUa |
||| ||||||||||| |
gaagCCCAGAGAGAACUGGAc |
|
|
BX890604.1 |
ucggggGAGUCU--CUUGACCUa |
|| ||| |||||||| |
gagaugCUGAGAAGGAACUGGAg |
|
|
LINC01204 |
ucggGGGAGUCUCUUGACCUa |
|| ||| ||||||| |
cugaCCAUCA-CUAACUGGAu |
|
|
JPX |
ucgggggagUC-UC-UUGACCUa |
|| || ||||||| |
ucaaaggaaAGAAGCAACUGGAa |
|
Для miR-130b предсказано взаимодействие с 21 lncRNA, для miR-149 – с 90 lncRNA, для miR-1249 – с 6 lncRNA и для miR-5195 – с 33 lncRNA. Из этого перечня длинных некодирующих РНК 24 взаимодействовали с несколькими микроРНК дифференциально экспрессирующимися у больных с разной эффективностью ЛТ (табл. 5, рис. 5).
Дальнейшим этапом работы стал анализ представленности транскриптов 24 lncRNA в биологических образцах двух групп больных РПК.
Таблица 5
Перечень длинных некодирующих РНК 24, взаимодействующих с несколькими микроРНК, дифференциально экспрессирующимися у больных с разной эффективностью ЛТ
|
lncRNA |
Число микроРНК взаимодействующих с lncRNA |
МикроРНК |
|
XIST |
4 |
hsa-miR-130b-3p, hsa-miR-149-5p, hsa-miR-195-5p, hsa-miR-1249-3p |
|
HELLPAR |
3 |
hsa-miR-149-5p, hsa-miR-195-5p, hsa-miR-5195-3p |
|
NEAT1 |
3 |
hsa-miR-130b-3p, hsa-miR-149-5p, hsa-miR-195-5p |
|
AC005537.1 |
2 |
hsa-miR-149-5p, hsa-miR-195-5p |
|
AC008124.1 |
2 |
hsa-miR-195-5p, hsa-miR-5195-3p |
|
AC016717.2 |
2 |
hsa-miR-130b-3p, hsa-miR-195-5p |
|
AC093297.2 |
2 |
hsa-miR-195-5p, hsa-miR-5195-3p |
|
AC097634.1 |
2 |
hsa-miR-195-5p, hsa-miR-5195-3p |
|
BX890604.1 |
2 |
hsa-miR-195-5p, hsa-miR-5195-3p |
|
CASC9 |
2 |
hsa-miR-195-5p, hsa-miR-5195-3p |
|
IQCH-AS1 |
2 |
hsa-miR-195-5p, hsa-miR-5195-3p |
|
LINC00662 |
2 |
hsa-miR-195-5p, hsa-miR-5195-3p |
|
LINC00707 |
2 |
hsa-miR-195-5p, hsa-miR-5195-3p |
|
LINC00943 |
2 |
hsa-miR-149-5p, hsa-miR-195-5p |
|
LINC01089 |
2 |
hsa-miR-130b-3p, hsa-miR-5195-3p |
|
LINC01468 |
2 |
hsa-miR-149-5p, hsa-miR-5195-3p |
|
LINC01547 |
2 |
hsa-miR-195-5p, hsa-miR-1249-3p |
|
MALAT1 |
2 |
hsa-miR-149-5p, hsa-miR-5195-3p |
|
MEG3 |
2 |
hsa-miR-195-5p, hsa-miR-5195-3p |
|
MIR503HG |
2 |
hsa-miR-149-5p, hsa-miR-195-5p |
|
MUC20-OT1 |
2 |
hsa-miR-149-5p, hsa-miR-5195-3p |
|
PVT1 |
2 |
hsa-miR-195-5p, hsa-miR-5195-3p |
|
RP1-178F10.3 |
2 |
hsa-miR-149-5p, hsa-miR-195-5p |
|
VASH1-AS1 |
2 |
hsa-miR-130b-3p, hsa-miR-149-5p |
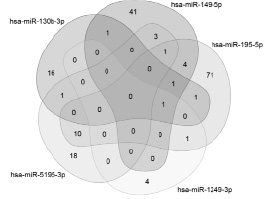
Рис. 5. Диаграмма Эйлера – Венна перекрывающихся взаимодействий 5 микроРНК и 202 длинных некодирующих РНК
Особенности экспрессии lncRNA и эффективность лучевой терапии. Анализ относительной представленности транскриптов длинных некодирующих РНК показал дифференциальные отличия по этому показателю двух групп пациентов с разным ответом на ЛТ. Так, у больных с полным ответом на лучевую терапию обнаружено статистически значимое (р < 0,05) снижение представленности транскриптов lncRNA XIST, HELLPAR, NEAT1, AC008124.1 и AC016717.2 в 2,5; 3,3; 2,0; 2,0 и 5,0 раз соответственно относительно нормальной ткани, а у больных с частичным ответом/ отсутствием ответа на лучевую терапию обнаружено статистически значимое (р < 0,05) увеличение представленности транскриптов lncRNA XIST, HELLPAR, NEAT1, AC008124.1, LINC01089, LINC01547 и VASH1-AS1 в 1,9; 2,9; 3,5; 3,7; 2,6; 4,7 и 1,9 раза соответственно относительно нормальной ткани. При этом между двумя группами пациентов по уровню представленности транскриптов lncRNA XIST, HELLPAR, NEAT1, AC008124.1, AC016717.2, LINC01089, LINC01547 и VASH1-AS1 также наблюдались статистически значимые отличия (р < 0,05) в 4,8; 9,7; 7,0; 7,4; 5,0; 3,1; 4,8 и 2,2 раза соответственно (рис. 6). Таким образом, полученные данные по представленности транскриптов длинных некодирующих РНК позволяют смоделировать сеть молекулярных взаимодействий, лежащих в основе радиорезистентности опухолевых клеток.
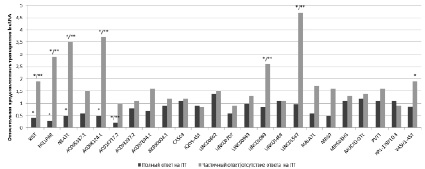
Рис. 6. Относительная представленность (относительно нормальной ткани) транскриптов длинных некодирующих РНК в опухолевой ткани прямой кишки двух групп больных с полным или частичным ответом / отсутствием ответа на ЛТ. * – статистически значимые отличия относительно нормальной ткани (р < 0,05), ** – межгрупповые статистически значимые отличия (р < 0,05)
Нарушения в регуляторной сети конкурентно-взаимодействующих РНК. Предыдущие этапы исследования позволили нам собрать необходимую информацию по всем компонентам регуляторной сети конкурентно-взаимодействующих эндогенных РНК (ceRNA), включая длинные некодирующие РНК (lncRNA), микроРНК (miRNA) и мРНК (экспрессия генов).
Комплексный анализ данных, представленных выше, позволил нам сформировать следующую модель (рис. 7). Снижение представленности транскриптов lncRNA XIST, HELLPAR, NEAT1, AC008124.1 и AC016717.2 у больных с полным ответом на лучевую терапию приводит к снижению эффективности взаимодействия lncRNA и таргетирующих их микроРНК (hsa-miR-130b-3p, hsa-miR-149-5p, hsa-miR-195-5p, hsa-miR-1249-3p, hsa-miR-5195-3p). Нарушение образования комплекса lncRNA-микроРНК приводит к увеличению количества свободных микроРНК hsa-miR-130b-3p, hsa-miR-149-5p, hsa-miR-195-5p и hsa-miR-5195-3p и их эффективному взаимодействию с генами-мишенями (мРНК). Соответственно снижается представленность транскриптов генов-мишеней микроРНК hsa-miR-130b-3p, hsa-miR-149-5p, hsa-miR-195-5p – RBBP8 и miR-5195-3p – H2AX, а следовательно, происходит снижение эффективности репарации ДНК.
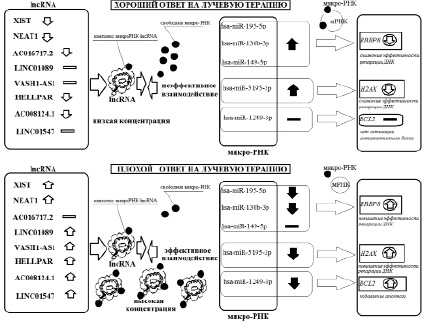
Рис. 7. Особенности регуляторной сети конкурентно-взаимодействующих РНК у больных с разным ответом на ЛТ
Противоположный эффект наблюдается у больных с плохим ответом на ЛТ. Увеличение представленности транскриптов lncRNA XIST, HELLPAR, NEAT1, AC008124.1, LINC01089, LINC01547 и VASH1-AS1 у этих больных приводит к увеличению эффективности взаимодействия lncRNA и микроРНК (hsa-miR-130b-3p, hsa-miR-149-5p, hsa-miR-195-5p, hsa-miR-1249-3p, hsa-miR-5195-3p). Образование комплекса lncRNA-микроРНК приводит к снижению количества свободных микроРНК hsa-miR-130b-3p, hsa-miR-149-5p, hsa-miR-195-5p и hsa-miR-5195-3p и снижению их взаимодействия с генами-мишенями (мРНК).
Соответственно увеличивается представленность транскриптов генов-мишеней микроРНК hsa-miR-130b-3p, hsa-miR-149-5p, hsa-miR-195-5p – RBBP8 и miR-5195-3p – H2AX, а следовательно, происходит увеличение эффективности репарации ДНК и формирование радиорезистентности (рис. 7).
Заключение
Комплексный анализ особенностей регуляторной сети конкурентно-взаимодействующих РНК и эффективности лучевой терапии опухолей прямой кишки позволил установить как механизмы формирования радиорезистентности, так и её предикторы. Эффективность лучевой терапии злокачественных опухолей прямой кишки связана с уровнем микроРНК (miR-195; miR-1273h; miR-6737; miR-6808; miR-3202; miR-5195; miR-4257; miR-5187; miR-149; miR-138; miR-6798; miR-6819; miR-4728; miR-1249; miR-557; miR-130b) и lncRNA (XIST, HELLPAR, NEAT1, AC008124.1, LINC01089, LINC01547 и VASH1-AS1), обеспечивающих эффективную регуляцию систем репарации ДНК (H2AX и RBBP-8) и апоптоза (BCL-2).
Исследование выполнено в рамках госзадания «Поиск предикторов радиорезистентности рака прямой кишки и разработка персонифицированных неоадъювантных терапевтических подходов».

