Микроорганизмы рода Lactobacillus широко распространены в природе, а некоторые виды являются важнейшими представителями микробиоты организма человека [1]. Лактобациллы длительное время привлекают внимание ученых – биохимиков, микробиологов, медиков, экологов, ввиду их потенциального значения для поддержания гомеостаза системы «человек-окружающая среда», сохранения здоровья населения, профилактики и лечения многих заболеваний различной этиологии. Актуальным вопросом является получение новых знаний о биологических свойствах и молекулярно-генетической структуре лактобацилл; создание новых пробиотических препаратов на их основе с помощью разных методических подходов к культивированию [2].
Главным конечным продуктом метаболизма лактобацилл является D- и L-молочная кислота. У гомоферментативных лактобацилл лактат составляет 90 % всех продуктов брожения. У представителей гетероферментативных видов в качестве конечных продуктов также образуется молочная кислота и углекислый газ [3]. Благодаря продукции органических кислот, перекисей и бактериоцинов многие штаммы лактобацилл проявляют выраженную антагонистическую активность в отношении патогенных микроорганизмов [4]. Биохимические и морфологические свойства лактобацилл являются в настоящее время основным и единственным критерием межродовой и видовой идентификации этих микроорганизмов. Однако при идентификации лактобацилл только с использованием стандартных микробиологических тестов могут возникнуть трудности. Так, например, идентификация близкородственных видов на основании физиолого-биохимических свойств затруднена вследствие общности свойств [5]. Идентификация молочнокислых бактерий только на основании морфологических, культуральных, физиолого-биохимических признаков в настоящее время является недостаточной, поскольку под воздействием различных факторов многие виды обладают высоким уровнем фенотипической изменчивости [6]. Молекулярно-генетические методы идентификации зарекомендовали себя как надежные и независящие от внешних факторов [7]. Целью настоящей работы являлась идентификация генетически близких видов лактобацилл, выделенных из желудочного кишечного тракта здоровых людей, с использованием анализа нуклеотидной последовательности гена 16S rRNA.
Материалы и методы исследования
Штаммы бактерий и методы культивирования. Объектами исследования являлись молочнокислые бактерии рода Lactobacillus, зарегистрированные в коллекции промышленных микроорганизмов «Института микробиологии и вирусологии» и по паспортным данным принадлежащих к Lactobacillus plantarum №22 и Lactobacillus cellobiosus №20. Культуры выращивали на среде MRS (Hi-Media, India), в течение 2-х суток при 35°С.
Определение и анализ нуклеотидных последовательностей генов 16SrRNA. Геномную ДНК выделяли методом Kate Wilson [11]. Концентрацию ДНК измеряли спектрофотометрическим методом с использованием спектрофотометра Nano Drop 1000 при длине волны 260 нм, а также проводили качественную оценку ДНК электрофоретическим методом.
Матрицы для секвенирования синтезировали с помощью ПЦР, используя универсальные праймеры 8f-5’–AgAgTTTgATCCTggCTCAg-3 и 806R-5’- ggACTACCAgggTATCTAAT-3 [12], что позволяло амплифицировать ген 16S rRNA почти полностью. Реакционная смесь (30 мкл) содержала 3 мкл 10х реакционного буфера (Fermentas), 2,5 мM MgCl2, 0,2 мM каждого дезоксирибонуклеозидтрифосфата (дНТФ), по 10 пмоль каждого из праймеров, 1единицу Taq –полимеразы Maxima Hot Start Taq DNA Polymerase (Fermentas) и 150 нг геномной ДНК в качестве матрицы.
ПЦР проводили в термоциклере Mastercycler pro S (Eppendorf). Реакцию начинали инкубированием смеси при 95 °С в течение 7 минут, затем следовало 30 циклов, состоящих из инкубаций: 95 °С – 30 секунд, 55 °С – 40 секунд , 72 °С – 1 мин. Завершающую элонгацию проводили при 72 °С в течение 10 минут.
Продукты амплификации разделяли в 1,5 % агарозном геле. Гели окрашивали этидиум бромидом. Электрофорез проводили в камере для горизонтального электрофореза Bio-RAD Basic, и источником тока «Consort EV-243». В качестве электродного буфера использовали 1хТАЕ-буфер. Документирование полученных результатов проводили, используя систему документаций гелей Gel Doc. Размеры молекул анализируемых образцов ДНК определяли путем сопоставления их электрофоретической подвижности маркеров – фрагмент ДНК известной молекулярной массы. В качестве маркера молекулярных масс использовали O’GeneRuler™ 100bp Plus DNA Ladder производства (Fermentas).
ПЦР продукты очищали от остатков олигонуклеотидов методом дефосфолирирования с помощью щелочной фосфатазы (SAP-shimp alkaline phosphatase) и эндонуклеазы I [13].
Секвенирование фрагментов гена 16S rRNA идентифицируемых бактерий проводили на автоматическом секвенаторе 3730хl DNA Analyzer (Applide Biosystems, США) с использованием набора Big Dye Terminator v3.1 Cycle Sequencing Kit (Applide Biosystems, США), придерживаясь рекомендаций производителя. Результаты секвенирования обрабатывали в программе SeqMan (Applide Biosystems). Поиск гомологичных нуклеотидных последовательностей генов 16S rRNA осуществляли с помощью программы BLAST (Basic Local Alignment Search Tool) в базе данных Gene Bank Национального центра биотехнологической информации США (htpp://www.ncbi. nlm. nih.gov). Идентификация была осуществлена относительно инвентарных номеров Genе Bank первых трех нуклеотидных последовательностей имеющих максимальное совпадение. Филогенетический анализ проводили с использованием программного обеспечения MEGA4 [14]. Выравнивание нуклеотидных последовательностей проводили, используя алгоритм ClustalW [15]. Для построения филогенетических деревьев использовали метод «объединения соседей» Neiighbor-Joining (NJ) [16]. Дополнительно в анализ были включены нуклеотидные последовательности близкородственных видов филогенетической группы Lactobacillus buchneri и Lactobacillus plantarum, депонированных в Международных базах данных.
Результаты исследования и их обсуждение
Филогенетический анализ нуклеотидных последовательностей генов 16S rRNA. В нашем исследовании генетическая идентификация 2-х штаммов была осуществлена методом определения прямой нуклеотидной последовательности фрагмента 16S rRNA гена, с последующим сравнением нуклеотидной идентичности с последовательностями, депонированными в международной базе данных Gene Bank, а также построением филогенетических деревьев с нуклеотидными последовательностями референтных штаммов.
В результате выделения ДНК были получены образцы с высокой концентрацией ДНК от 80 до 150 нг/мкл, соотношение длин волн 260/280 в среднем составило 2,12. Методом ПЦР был амплифицирован фрагмент 16S rRNA гена, молекулярной массой размером около 800 п.н. Результаты амплификации образцов с отрицательным контролем отображены на рис. 1.
Продукты ПЦР амплификации были использованы для определения нуклеотидной последовательности.
Оценка нуклеотидной идентичности анализируемых последовательностей фрагмента 16S rRNA гена с референтными последовательностями филогенетически близких видов представлена в таблице.
Для исключения методической погрешности анализа 16S rRNA гена [17], дополнительно было проведено построение филогенетических деревьев (рис. 2, 3).
После удаления последовательности праймеров в неперекрывающихся областях были получены нуклеотидные последовательности размером 650 п.н. При выравнивании было выявлено, что нуклеотидные последовательности фрагмента 16S rRNA гена референтных штаммов Lactobacillus farraginis (Gene Bank AB690214.1), Lactobacillus hilgardii (Gene Bank KC351904.1), Lactobacillus pentosus (АВ362673), Lactobacillus plantarum (AJ640078), имели размер 560 и 550 п.н. соответственно. В связи с этим, для формирования однородной выборки размером 550 п.н. была удалена часть нуклеотидных последовательностей у исследуемых штаммов.
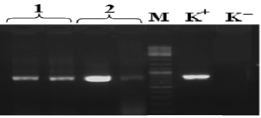
Рис. 1. Электрофорерограмма ПЦР – продуктов, полученных при амплификации фрагмента гена 16S rRNA: 1 – штамм №22, 2 – штамм №20, (в двух повторностях), М – маркер молекулярной массы O’GeneRuler™ 100bp Plus DNA (Fermentas) (100 -1000 п.н. шаг 100 п.н.), (К-) – отрицательный контроль постановки ПЦР, (К+) – положительный контроль постановки ПЦР
Результаты идентификации нуклеотидной последовательности в международной базе данных
|
Наименование штамма |
Инвентарный номер GeneBank |
Наименование штамма |
% совпадения |
|
Lactobacillus plantarum №22 |
AB690214.1 |
Lactobacillus farraginis |
100 |
|
KC351904.1 |
Lactobacillus hilgardii |
98 |
|
|
Lactobacillus cellobiosus №20 |
АВ362673 |
Lactobacillus pentosus. |
100 |
|
AJ640078 |
Lactobacillus plantarum |
98 |
Процент идентичности фрагментов нуклеотидных последовательностей 16S rRNA гена между Lactobacillus plantarum №22 и Lactobacillus farraginis составил 100 %, а между Lactobacillus cellobiosus №20 и Lactobacillus pentosus также составил 100 %. Нуклеотидные последовательности гена 16S rRNA двух идентифицируемых культур Lactobacillus plantarum №22, Lactobacillus cellobiosus №20 расположены на одной ветви с референтными штаммами Lactobacillus farraginis и Lactobacillus pentosus.
При идентификации в Genе Bank данных культур по 16S rRNA максимальная идентичность была получена с референтными штаммами Lactobacillus farraginis и Lactobacillus pentosus. Данные культуры по гену 16S rRNA были идентифицированы как Lactobacillus farraginis и Lactobacillus pentosus.
Результаты филогенетического анализа последовательностей гена 16S rRNA у изучаемых штаммов представлены на филогенетическом дереве (рис. 2-3), построенном в программе MEGA4, с использованием Neiighbor-Joining кластерного метода расчета генетических расстояний и bootstrap анализа, отражающего достоверность кластеризации.
Как видно из представленных данных, по характеру последовательностей штаммы можно распределить в 2 кластера, один из которых составляет культуру Lactobacillus farraginis, нуклеотидные последовательности которых характеризуются 100 % сходством. Второй кластер включает штамм Lactobacillus hilgardii сходство последовательностей гена 16S rRNA у которого составило 98 % (рис. 2).
Как видно из рис. 2, нуклеотидные последовательности штамма идентифицируемого как Lactobacillus plantarum № 22, при идентификации в Genе Bank находятся на одной филогенетической ветви с Lactobacillus farraginis. Таким образом, штамм Lactobacillus plantarum № 22 на основании анализа нуклеотидной последовательности гена 16S rRNA идентифицирован как Lactobacillus farraginis. В основном кластере бактерий Lactobacillus farraginis, характеризовался более высоким уровнем вариабельности генов 16S rRNA, чем кластер, образуемый Lactobacillus hilgardii. Штамм Lactobacillus plantarum №22 был тесно связан с референтным штаммом Lactobacillus farraginis, что позволяет рассматривать его как филогенетически наиболее близкий к типовому штамму вида Lactobacillus farraginis. Это является обоснованием для правомерности его отнесения к Lactobacillus farraginis (рис. 3).
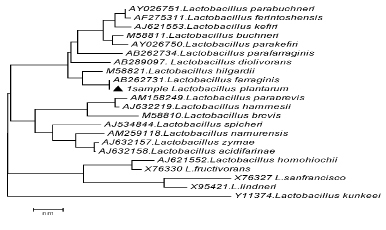
Рис. 2. Филогенетическое дерево, построенное на основании анализа фрагмента гена 16S rRNA, показывающее родство группы Lactobacillus buchneri
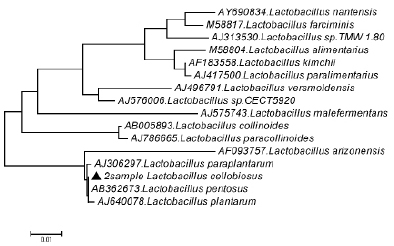
Рис. 3. Филогенетическое дерево, построенное на основании анализа фрагмента гена 16S rRNA показывающее родство группы Lactobacillus plantarum
Как видно на рис. 3, при филогенетическом анализе фрагмента 16S rRNA гена штамм Lactobacillus cellobiosus № 20 был объединен в один кластер с Lactobacillus paraplantarum. При идентификации в Genе Bank штамм Lactobacillus cellobiosus № 20 находится на одной филогенетической ветви образуемой бактериями Lactobacillus pentosus и Lactobacillus plantarum, что указывало на его принадлежность к одному из этих видов и исключало возможность отнесения к Lactobacillus cellobiosus. Разграничение видов Lactobacillus pentosus и Lactobacillus plantarum на основании результатов проведенного филогенетического анализа оказалось невозможным.
Причиной этого можно считать низкую вариабельность нуклеотидных последовательностей генов 16S rRNA. По данным Genе Bank уровень генетического родства 16S rRNA гена исследуемой культуры Lactobacillus cellobiosus № 20 по отношению к Lactobacillus pentosus составил – 100 %, а к наиболее близкородственному виду Lactobacillus plantarum – 98 %. При анализе филогенетического дерева видно, что идентифицируемая культура Lactobacillus cellobiosus №20 филогенетически связана с Lactobacillus pentosus и Lactobacillus plantarum. Важно отметить, что нуклеотидная идентичность между филогенетически близкородственными бактериями группы Lactobacillus plantarum, как Lactobacillus pentosus и Lactobacillus plantarum составила более 98 %, что не позволяет однозначно дифференцировать указанные виды.
Таким образом, в данном исследовании представлены результаты молекулярно-генетической идентификации двух штаммов молочнокислых бактерий на основе анализа нуклеотидных последовательностей 16S rRNA гена, выделенных из желудочно-кишечного тракта здоровых людей. Оба штамма молочнокислых бактерий были просеквенированы и проанализированы. Сравнительный анализ нуклеотидных последовательности обоих штаммов молочнокислых бактерии показал их высокую идентичность.
Коллекционные штаммы лактобацилл по результатам нашего анализа последовательностей их генов 16S rRNA были разделены на 2 группы. В первую группу Lactobacillus buchneri вошел штамм Lactobacillus plantarum №22, видовая принадлежность которого не соответствует их исходным паспортным данным, составленным на основании традиционных микробиологических методов исследования. Ко второй группе Lactobacillus plantarum отнесен штамм Lactobacillus cellobiosus №20, у которого последовательность гена 16S rRNA также не соответствует видовому положению, указанному в паспортных данных. Сравнение нуклеотидной последовательности генов 16SrRNA коллекционных штаммов с таковой международных баз данных позволили установить, что штаммы №22 и №20, идентифицированные ранее, как штаммы Lactobacillus plantarum №22 и Lactobacillus cellobiosus №20, на самом деле относятся к Lactobacillus farraginis и Lactobacillus pentosus. Секвенирование генов 16S rRNA выявило высокий уровень гомологии с представителями рода Lactobacillus: штамм Lactobacillus plantarum № 22 – 100 % с Lactobacillus farraginis; а штамм Lactobacillus cellobiosus № 20 – 100 % с Lactobacillus pentosus. Таким образом, более целесообразно проводить генетическую идентификацию на основе анализа нуклеотидной последовательности 16S rRNA гена, так как она позволяет идентифицировать культуры до видов и сократить время исследований.
Авторы выражают признательность сотрудникам Национальной научной лаборатории биотехнологии коллективного пользования РГП «Национальный центр биотехнологии Республики Казахстан» КН МОН РК города Астаны – зав. лаборатории Е.В. Жолдыбаевой, к.б.н. А.Б. Шевцову, мнс. Б.Т. Байкаре за консультации при написании статьи.
Работа выполнена в рамках проекта «Обеспечение сохранения и развития коллекционных фондов растений, животных, микроорганизмов, вирусов и уникальных генетических банков как основы фундаментальных исследований» в рамках программы Грантового финансирования МОН РК на 2012-2014 гг.

