Количественная ПЦР в режиме реального времени является чувствительным, специфичным и простым методом для анализа экспрессии генов c помощью которого можно точно и быстро определять изменения уровня экспрессии интересующего гена в различных биологических образцах.
Одним из вариантов количественного анализа содержания транскриптов является их оценка и нормирование по отношению к референсным генам, которые амплифицируются в ПЦР одновременно с исследуемыми генами. Выбор наиболее стабильных референсных генов является важной задачей, так как по ним проводится нормирование данных, и недостаточная стабильность этих генов может существенно искажать результаты количественной ПЦР [7].
Вариабельность экспрессии любых референсных генов не является постоянной и в различных экспериментальных условиях может сильно изменяться, поэтому выбор генов для нормализации данных является необходимым этапом эксперимента, и даже применение широко используемых в современных исследованиях референсных генов требует предварительной оценки их стабильности [9].
Данных об использовании референсных генов губок крайне мало. В единичных исследованиях морских и пресноводных губок в количественной ПЦР для нормализации данных использовались такие гены как α-тубулин, 28S рРНК, убиквитин [6].
Проблема выбора генов часто усложняется в случае нетиповых моделей биологических экспериментов, так как для многих организмов недостаточно информации о геноме исследуемого объекта. К таким малоисследованным объектам относятся пресноводные эндемичные байкальские губки. Наблюдающаяся в настоящее время массовая гибель байкальских губок, и в первую очередь ветвистой формы Lubomirskia baicalensis, требует срочной оценки их состояния, в том числе с помощью анализа экспрессии генов с применением ПЦР в режиме реального времени. Кроме того, исследования различных биологических процессов в байкальских губках представляют большой теоретический и практический интерес в связи с уникальностью этих древних организмов и возможностью их использования в качестве различных моделей, в частности, для изучения процессов биосилификации [2].
Таким образом, целью данной работы является выбор референсных генов, для нормирования данных количественной ПЦР. Для этого нами проанализированы шесть потенциальных референсных генов: рибосомальный белок 11 (rpl11), β-актин (actb), эукариотический фактор элонгации трансляции 1α (eef1A1), миозин II (myo), лектин L6 (lgalsl), профилин (pfn), и проведена оценка стабильности экспрессии каждого из них с помощью программ NormFinder и BestKeeper.
Материалы и методы исследования
Взрослые ветвистые L. baiсalensis собирали при помощи водолазов в южной котловине Байкала. Живые образцы губок транспортировали в лабораторию при температуре 3-4 °С. Видовую идентификацию губок проводили по внешним морфологическим признакам. Для выбора референсных генов анализировали 9 образцов губок и 8 образцов примморф. Взрослые губки были собраны в феврале 2013, марте 2012, апреле 2013, июне 2013 и августе 2014 на глубине 10 м. Примморфы получали из взрослых губок и после двух месяцев культивирования индуцировали дабавлением силиката натрия (Na2SiO3•9H2O) до конечной концентрации 70 μМ. Примморфы размером 3 мм в диаметре из неиндуцированной и индуцированных три, пять и семь дней культур отбирали в количестве 10 штук для экстракции РНК. Выделение суммарной РНК проводили с помощью реагента Trisol LS (“Ambion/Life Technologies”) по протоколу производителя. Концентрацию РНК измеряли на спектрофотометре Nanovue (GE Healthcare) и 1 мкг использовали для обработки свободной от РНКаз ДНКазой 1 (“Thermo Scientific”), которую брали из расчета 1 ед на 1 мкг РНК и инкубировали при 37 °С в течение 15 мин в 1x буфере для ДНКазы 1, содержащем Mg2+, инактивировали фермент при 65 °С 10 мин и половину объема (5 мкл) использовали для постановки реакции обратной транскрипции (ОТ). Качество РНК контролировали с помощью электрофореза при окрашивании РНК бромистым этидием. При наличии двух четких полос рРНК, одинаковой интенсивности окраски и отсутствии низкомолекулярной РНК, пробы РНК использовали для постановки ОТ и ПЦР. Реакцию ОТ проводили в 20 мкл реакционной смеси с помощью набора для обратной транскрипции MMLV RT kit (Евроген) и гексаолигонуклеотидными праймерами, согласно инструкции, реакционную смесь инкубировали в течение часа при 37 °С.
Последовательности праймеров для амплификации генов actb, eef1A1, rpl11 выбраны на основе выравнивания (из имеющихся в базе данных GenBank нуклеотидных последовательностей морских и пресноводных губок JX050219, HQ677599, HQ693078, JQ606685, FJ529210, DQ087461, AY580191, XM_003384761, GQ330985, GQ330974, HF570277). Выравнивание последовательностей и выбор праймеров проводили в программах BioEdit (версия 7.0.9.0) [3] и OligoAnalyzer 3.1 (http://eu.idtdna.com). Полученные в ПЦР ампликоны ожидаемой длины вырезали из геля 0,8% агарозы, очищали и клонировали в векторе pAL-TA (“Евроген”), согласно инструкции производителя. Клоны, содержащие вставки, секвенировали на приборе ABI Prism 3130xl Genetic Analyzer (“Applied Biosystems”). Специфичность полученных нуклеотидных последовательности оценивали в программе BLAST (http://blast.ncbi.nlm.nih.gov/Blast.cgi) и с помощью анализа последовательностей искомых генов губок, в том числе в сравнении с последовательностями мРНК близкородственной пресноводной губки E. muelleri (номер проекта в Genbank PRJNA230473). Последовательности трех других генов, myo (миозин), lgalsl (лектин), pfn (профилин) выбраны на основе данных частичного секвенирования транскриптома L. baiсalensis. Полученные нуклеотидные последовательности мРНК исследуемых генов депонированы в базу данных GenBank с номерами доступа KR708639 – KR708644.
На основе полученных последовательностей с помощью программы Primer3Plus [10] были выбраны праймеры для проведения количественной ПЦР. Специфичность праймеров оценена с помощью программы BLAST и выбраны праймеры, специфичные исследуемому объекту и не способные амплифицировать гены симбионтов губок, бактерий и зеленых водорослей. Для проведения количественной ПЦР в реальном режиме времени исопльзовали следующие праймеры:
rpl11_f 5`-ATGAATTGCGCAAGGAAAAC-3`, rpl11_r 5`-CTTGTTCACACGGTGTTTCG-3` (203 п.н.); actb_f 5`-ACTGGGACGACATGGAGAAG-3`, actb_r 5`-TGGCTAGGGTGTTGAAGGTC-3` (162 п.н.); eef1A1_f 5`-GCAGCTAATCGTTGGTGTCA-3`, eef1A1_r 5`-GTAGGTCGGTGCTTCTCTCG-3` (182 п.н.); myo_f 5`-GAGCAGGGTACAATGGAGGA-3`, myo_r 5`-TCAATGCCAGTCAACAGAGC-3` (155 п.н.); lgalsl_f 5`-CCCATCCACAGGCAGAGTAT-3`, lgalsl_r 5`-GGCTACATCTGGGGAGTCAA-3` (168 п.н.); pfn_f 5`-CCAAGCTTCTGGCATCTACC-3`, pfn_r 5`-CTTGTTCGCATTTCCCTGTT-3` (175 п.н.).
Реакцию количественной ПЦР проводили в 0,2 мл пробирках на приборе Rotor-Gene Q (“Qiagen”) для чего использовали готовую 5x смесь qPCRmix-HS (Евроген, Россия), содержащую интеркалирующий краситель SYBR Green I, 5 пмоль каждого праймера, 1 мкл матрицы кДНК, воду Milli-Q до конечного объема 15 мкл. Для проведения количественной ПЦР и анализа экспрессии гена силикатеина альфа (sila) использовали праймеры silAf 5`-GGAGATTGTGGTGCCAGCTATGC-3` и silAr 5`-GCTCTCTGTGTCGATTCCACCGT-3` (204 п.н.), выбранные на основе последовательности с номером AJ786771 в базе данных GenBank.
Каждый образец амплифицировали в трех повторностях в режиме 4 мин 95 °С, с последующими 40 циклами: 15 с 95°С, 15 с – 61 °С, 20 с – 72°С. По окончании амплификации анализировали кривые плавления в диапазоне температур 61 – 95°С, флуоресценцию измеряли с шагом 0,5°С. В качестве отрицательных контролей использовали реакционную смесь без матрицы и с РНК матрицей без этапа обратной транскрипции для контроля контаминации геномной ДНК. Значения порогового цикла (Ct) рассчитывали как среднее значение трех повторностей каждого образца.
Оценку эффективности ПЦР проводили при амплификации каждого гена, анализируя по 6 двукратных разведений кДНК после построения стандартных кривых с помощью программного обеспечения прибора Rotor-Gene Q (“Corbett Research”). Анализ стабильности экспрессии генов проводили с помощью программ NormFinder и BestKeeper, имеющихся в свободном доступе [1, 8]. Расчет количества кДНК кандидатных референсных генов проводили с помощью метода дельта Сt [5].
Результаты исследования и их обсуждение
Для всех иccледуемых в качестве референсных генов, rpl11, actb, eef1A1, myo, lgalsl и pfn, получены специфичные нуклеотидные последовательности и разработаны праймеры для количественной ПЦР. Предварительная оценка показала, что эффективность реакций варьировала от 88 до 107%, а коэффициенты корреляции были выше 0,99, что позволяет применять метод ΔΔСt для определения уровня экспрессии генов [5]. Кривые плавления для всех реакций показывали один пик, соответствующий температуре плавления специфичного ампликона. Анализ пороговых значений Ct, показал, что в примморфах и взрослых губках L. baicalensis представленность транскриптов в образцах уменьшалась в ряду actb>pfn>eef1A1>rpl11>myo>lgalsl. Пороговые значения Ct исследованных генов варьировали в диапазоне от 11 до 22 циклов.
Для поиска наиболее стабильно экспрессирующихся генов проводили количественную ПЦР и оценивали пороговые значения Сt в группах примморф и взрослых губок. Полученные результаты анализировали с помощью программ BestKeeper и NormFinder, использующих различные алгоритмы для выбора наиболее стабильных генов (таблица).
Значения, полученные в программах BestKeeper и NormFinder для потенциальных референсных генов
|
Примморфы |
actb |
rpl11 |
myo |
eef1A1 |
lgalsl |
pfn1 |
||
|
BestKeeper |
SD |
0,97 |
0,99 |
0,88 |
1,21 |
0,98 |
0,95 |
|
|
CV |
6,89 |
5,11 |
4,26 |
6,61 |
4,73 |
5,31 |
||
|
r |
0,987 |
0,901 |
0,870 |
0,691 |
0,691 |
0,989 |
||
|
p |
0,001 |
0,002 |
0,005 |
0,001 |
0,058 |
0,001 |
||
|
NormFinder |
Intragroup variation |
0,015 |
0,017 |
0,114 |
0,021 |
0,067 |
0,004 |
|
|
Intergroup variation |
0,093 |
-0,145 |
0,143 |
-0,060 |
0,201 |
-0,233 |
||
|
Губки |
BestKeeper |
SD |
0,94 |
0,85 |
0,70 |
0,94 |
1,02 |
0,83 |
|
CV |
7,59 |
4,89 |
3,75 |
5,86 |
5,23 |
5,62 |
||
|
r |
0,805 |
0,925 |
0,975 |
0,882 |
0,794 |
0,927 |
||
|
p |
0,005 |
0,001 |
0,001 |
0,001 |
0,006 |
0,001 |
||
|
NormFinder |
Intragroup variation |
0,206 |
0,229 |
0,043 |
0,398 |
0,527 |
0,619 |
|
|
Intergroup variation |
-0,093 |
0,145 |
-0,143 |
0,060 |
-0,201 |
0,233 |
||
|
Примморфы + губки |
Stability value |
0,199 |
0,244 |
0,239 |
0,203 |
0,326 |
0,324 |
R – коэффициент корреляции, CV – коэффициент вариации, SD – стандартное отклонение, p – значение вероятности, Intragroup variation – внутригрупповая вариация, Intergroup variation – межгрупповая вариация, Stability value – показатель стабильности (Normfinder). Жирным шрифтом приведены значения показателей генов не прошедших критерии отбора референсных генов.
Программа BestKeeper основывана на анализе пороговых значений Ct, показателях стандартного отклонения (SD) и коэффициента корреляции (R) для каждого гена в ряду данных. Гены с наиболее стабильной экспрессией имеют наименьшие показатели SD и максимально близкие к 1 показатели R, кроме того, гены с величиной SD>1, не соответствуют требованиям к референсным генам и исключаются из дальнейшего анализа. С помощью программы BestKeeper отдельно анализировали два набора данных, полученных для примморф и взрослых губок. В результате анализа наиболее стабильными генами с наименьшими показателями SD в обеих группах были гены myo и pfn, а наименее стабильными генами оказались гены lgalsl и eef1A1.
С помощью программы NormFinder были проанализированы те же данные количественной ПЦР, полученные для двух групп образцов, примморф и взрослых губок. Алгоритм анализа NormFinder основан на значениях Сt референсных генов, переведенных в линейную шкалу с помощью калибровочной кривой или метода дельта Ct (использованного в данной работе). Выбор наиболее стабильных генов обусловлен минимальными показателями стандартного отклонения внутри и между исследуемыми группами. В результате анализа референсных генов в группах примморф и взрослых губок программой был определен наиболее стабильный ген – actb и наилучшая комбинация двух генов – actb и rpl11. Несмотря на близкий к actb показатель стабильности, ген eef1A1 проявил существенную внутригрупповую вариабельность (в группе взрослых губок), что не позволяет рекомендовать его в качестве референсного. Ген myo показал наиболее близкие значения стабильности и вариабельности c геном rpl11. Гены pfn и lgalsl в программе Normfinder оказались самыми нестабильными в ряду анализируемых генов (показатель стабильности >0,3) и исключены из дальнейшего анализа.
Полученные с помощью двух программ результаты несколько различались, референсные гены, выбранные с помощью программы NormFinder – actb и rpl11, показали средние значения стабильности при анализе с помощью программы BestKeeper, однако наименее стабильным геном в обоих случаях был ген лектина – lgalsl. Отличия в результатах анализа с помощью программ BestKeeper и NormFinder объясняются различными алгоритмами для определения стабильности генов. По сравнению с BestKeeper, NormFinder, учитывающая внутри- и межгрупповую вариацию имеет преимущество при выборе стабильных генов в дух группах, в данном случае, взрослых губок и примморф. На основе обеих программ, согласно рекомендациям по использованию минимум трех генов для корректного нормирования данных количественной ПЦР [4], в качестве референсных нами выбраны гены actb, rpl11 и myo.
Для экспериментального подтверждения пригодности выбранных референсных генов была проведена оценка изменения экспрессии гена silα во взрослых губках, собранных в различные сезоны года (рисунок).
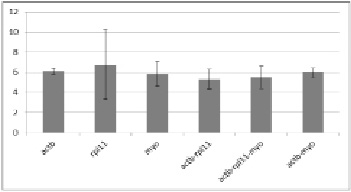
Относительное изменение уровня экспрессии гена silα в губках L. baicalensis в весенне-летний период.
Блоки показывают кратность изменения уровня silα (Fold Change) в губках, совбранных в апреле и июне по сравнению с губкой, собранной в феврале. На горизонтальной оси отмечены референсные гены и их комбинации, использованные для нормирования данных. Планки погрешностей показывают стандартное отклонение.
Ген silα является маркером склероцитов, клеток, продуцирующих спикулы. Предполагается, что уровень экспрессии гена silα увеличивается в весенний период, когда наблюдается активный прирост тела губки. Результаты исследования экспрессии silα в L. baicalensis получены с помощью метода ΔΔСt. В качестве референсных генов использованы как отдельные гены actb, rpl11, myo, так и их комбинации. Для оценки изменения экспрессии silα анализировали два образца губок, собранных в апреле и июне. Для сравнения использовали губку, собранную в феврале месяце, в период минимального роста. Как показано на рисунке, в весенне-летний период произошло увеличение уровня экспрессии silα в 5,5 раз при нормировании к комбинации генов actb, rpl11 и myo, что подтверждает ожидаемые результаты. При этом значения, полученные при использовании какого-либо одного из выбранных референсных генов, варьируют существеннее (0,95 Fold Change), чем при использовании двух и трех генов для нормализации, что подтверждает преимущество применения комбинации трех стабильных референсных генов.
Заключение
В результате работы нами проведен поиск и анализ референсных генов для нормализации данных количественной ПЦР в примморфах и взрослых байкальских губках L. baicalensis. Из шести проанализированных кандидатов с помощью программ NormFinder и Bestkeeper определены гены с наиболее стабильной экспрессией – actb, rpl11 и myo, тогда как гены eef1a1, lgalsl и pfn не соответствовали критериям стабильности референсных генов. Выбранные гены и их комбинации были использованы для нормирования данных при анализе экспрессии гена silα в губках L. baicalensis и показано, что в весенне-летний период уровень экспрессии увеличился в 5,5 раз. В результате оценки потенциальных референсных генов с помощью программ BestKeeper и NormFinder, а также экспериментального исследования экспрессии гена silα, для нормализации данных количественной ПЦР рекомендованы комбинации трех референсных генов: actb, rpl11 и myo. Выбранные референсные гены позволяют нормализовать данные количественной ПЦР для исследования экспрессии генов L. baicalensis в природной среде и в культуре примморф. Полученные результаты будут способствовать корректному анализу изменений, происходящих на генном уровне, как при исследовании процессов биосилификации, так и при воздействии различных факторов, в том числе в условиях стресса и массовой гибели байкальских губок.
Работа выполнена при поддержке проекта РФФИ № 13-04-00482.

