При идиопатическом сколиозе (ИС) происходит трехмерная деформация грудного и/или поясничного отделов позвоночника. При этом основная роль в деформации выпадает на торсию, т.е. на скручивание вокруг вертикальной оси. Если принимать искривление позвоночника (угол Кобба) по крайней мере, в 10˚, как идиопатический сколиоз, то он является частым заболеванием и встречается у 0,5–10 % подростков во всех человеческих популяциях [1]. Активно развиваться заболевание начинает обычно в период бурного роста в подростковом периоде, и выраженными факторами риска являются потенциал роста и принадлежность к женскому полу. Не доставляя пациентам явных неудобств, начальные стадии заболевания часто остаются незамеченными. Современная классификация определяет подростковый идиопатический сколиоз при диагностике заболевания в возрасте 11–20 лет [2]. Чаще всего заболевание диагностируется попутно в ходе медицинских осмотров по другим поводам. Идиопатическим сколиозом определяют все деформации позвоночника, развившиеся постнатально без видимых причин. Наследственная природа идиопатического сколиоза считается установленным фактом, и наследование заболевания является полигенным, хотя в литературе описаны семьи с Менделевским принципом наследования [3]. Исследования в разных популяциях показали, что у пациента с диагностированным идиопатическим сколиозом выявляются 11,1 % родственников 1-й степени родства, 2,4 % родственников 2-й степени родства, и 1,1 % родственников 3-й степени родства с деформациями позвоночника [4]. Резонным решением в такой ситуации был поиск молекулярных маркеров сколиоза, а при их наличии появится возможность выявлять эти маркеры в геноме тем или иным способом и рассчитывать риски развития заболевания у индивида.
В своих предыдущих публикациях [5–7] мы представили данные из литературных источников о связи ряда генов и мутаций в них с развитием и протеканием подростковых идиопатических сколиозов. Мутации являются не только причинами каких-либо заболеваний, но и могут быть молекулярными маркерами заболеваний, если они не изменяют важных функций генов или находятся в не кодирующих районах генома, при этом достоверно связаны с развитием заболевания. Это дает возможность использовать молекулярные маркеры для выявления генетической предрасположенности к заболеванию. Выявленных нами молекулярных маркеров сколиоза оказалось достаточно много, и это определило выбор ДНК-микрочиповой технологии, как наиболее подходящей, для анализа геномов на наличие таких маркеров. ДНК-микрочип позволит одновременно анализировать все размещенные на нем маркеры и получать информацию для расчетов риска развития сколиоза. Анализ позволит выявлять детей с риском развития у них сколиоза до клинических проявлений заболевания, в некоторых случаях определять характер его протекания и прогнозировать результаты его лечения. Дети с высокими рисками развития сколиоза будут определяться в группы пристального наблюдения и проходить частые медицинские освидетельствования для того, чтобы выявить прогресс в развитии заболевания и принять правильное и своевременное решение о выборе метода лечения – консервативного либо хирургического.
В Новосибирском НИИ травматологии и ортопедии им. Я.Л. Цивьяна разработан ДНК-микрочип, для которого был сформирован набор из 79 однонуклеотидных полиморфных локусов (ОНП) или, в английском варианте, «single nucleotide polymorphism» (SNP), для которых было показано, что они достоверно ассоциированы с идиопатическим сколиозом. Не все молекулярные маркеры (мутации) в этом списке являются по своей природе однонуклеотидными заменами, есть в нем и инсерции, и делеции, и дупликации, часто затрагивающие несколько нуклеотидов. По принятой классификации, все эти типы маркеров (мутаций) объединены в класс SNP, выявленный набор маркеров не является окончательным, он может быть со временем существенно изменен, поскольку технология легко позволяет добавлять или удалять маркеры с микрочипа. В набор были включены маркеры из 53 генов и районов хромосом, если полиморфные локусы располагались в межгенных промежутках. Практически все эти гены никак не связаны с развитием или функционированием соединительной ткани, а некоторые из них даже косвенно не вовлечены в генные каскады, обеспечивающие метаболизм соединительных тканей. К тому же практически все выбранные маркеры располагаются в некодирующих частях генов – интронах. Каким образом выявленные маркеры ассоциированы с заболеванием сколиозом, на сегодняшний день никто ответить не может. Не было выявлено ни одного основного гена, изменение функций которого наверняка приводят к развитию сколиоза. Авторы большинства публикаций, посвященных генетике сколиоза, пишут, что его наследование является полигенным, для которого список генов не определен. Из большого списка маркеров, для которых пытались выявить ассоциацию с заболеванием, нами были выбраны 79 маркеров для создания прогностической тест-системы, определяющей риски возникновения заболевания или особенности его протекания. Анализ геномов позволяет выявлять генетическую предрасположенность к сколиозам при скрининговых обследованиях населения либо на ранних стадиях заболевания по назначению врача, если в анамнезе пациента имеются случаи заболевания. Информация о генетических предпосылках заболевания поможет лечащему врачу выбрать адекватный метод лечения. Например, разработанный компанией Axial Biotech, Inc «ScoliScore™ AIS Prognostic Test» используется в ряде ортопедических клиник в США: «Virginia Spine Institute», «Schumacher Chiropractic Clinics», «Pedspine Clinic in Illinois Bone & Joint Institute» и помогает лечащим врачам выявить генетические причины сколиозов и выбрать стратегию лечения. В 2012 г. ведущий вертебрологический журнал «Spine» опубликовал результаты тестирования «ScoliScore™ AIS Prognostic Test», которые убедительно показали, что предсказание рисков развития заболевания и сложности его протекания, сделанные при помощи теста, намного точнее, чем сделанные классическими методами [8]. Позже коллектив тех же авторов провел другое исследование и показал, что «ScoliScore™ AIS Prognostic Test», не различал пациентов с прогрессией кривизны позвоночника и без нее, а отрицательные и положительные прогностические значения были ниже в этом исследовании, чем в ранее опубликованной валидации теста [9]. Однако дальнейшие исследования [10] показали, что этот тест хорошо работает только на американцах европейского происхождения и оказался малопригодным даже для канадцев с французскими корнями. Так же он не сработал на выборке пациентов из Японии [11], из Китая [12]. Это, по-видимому, обусловлено тем, что популяции из разных регионов мира характеризуются своими генетическими маркерами предрасположенности к сколиозам и к прогрессированию заболевания. Исследования специфических молекулярных маркеров сколиоза не проводились на территории России вообще и в Сибирском регионе в частности. Локальные популяции россиян могут быть по-своему уникальными. В Сибирско-Дальневосточном регионе проживают не только представители европеоидной расы, но и монголоиды: буряты, тувинцы, хакасы, якуты и другие восточноазиатские и североазиатские народы, а также выходцы из Средней Азии. Популяция европеоидов в Сибирском регионе тоже имеет свои особенности из-за смешивания с местными и среднеазиатскими монголоидами. Кроме того, сибирская популяция в целом может характеризоваться отличными от других популяций частотами встречаемости таких маркеров. Созданный ДНК-микрочип после масштабных испытаний позволит выявить генетические маркеры сколиоза, наиболее распространенные в регионе.
Цель исследования: создать экспериментальный образец ДНК-микрочипа для выявления генетической предрасположенности к заболеванию идиопатическим сколиозом.
Материалы и методы исследования
Последовательности олигонуклеотидов, содержащих однонуклеотидные полиморфизмы (маркеры), брались из геномной базы данных NCBI. Длина олигонуклеотида, рассчитывалась в зависимости от температуры плавления олигонуклеотида, и составляла от 19 до 45 нуклеотидов. Все олигонуклеотиды для печати на подложку уравнивались по температуре плавления равной 62 °С.
Синтез разработанных олигонуклеотидов был осуществлен в одной из компаний, предлагающих такую услугу. После синтеза олигонуклеотиды были разведены деионизированной водой до концентрации 100 нг/мкл. При подготовке к печати на подложку олигонуклеотиды разводились 1/1 буфером для печати от «Arrayit» Micro Spotting Solution 2X, кат. № MSS с конечной концентрацией 50 нг/мкл.
Печать олигонуклеотидов на стеклянные носители Super Epoxy 2, кат. № SME2 от «Arrayit» производилась на принтере SpotBot® Personal Microarrayer Complete кат. № SPA3XP от того же производителя. Каждый олигонуклеотид печатался в трех повторах с расстоянием между точками равным 175 микрометрам в строках и столбцах. Стеклянный носитель имел размеры 75х25х1 мм и был покрыт эпоксидным составом для лучшего сцепления олигонуклеотидов с поверхностью. После печати стекла с напечатанными микрочипами выдерживались в термостате при температуре 80 °С в течение 1 часа для лучшей фиксации олигонуклеотидов. Готовые микрочипы хранились в закрытых пластиковых коробках при комнатной температуре. Обозначения маркеров (однонуклеотидных полиморфных локусов) приводятся согласно международной номенклатуре, используемой в геномной базе данных NCBI.
Сбор образцов венозной крови (5 мл) проводился во время клинического обследования пациентов с согласия их родственников. Одновременно образцы крови брались у родственников при их согласии участвовать в исследовании. Кровь забиралась в вакуумные пробирки, содержащие ЕДТА для предотвращения свертывания крови. После доставки образцов крови в лабораторию пробирки с кровью центрифугировались для разделения цельной крови на фракции, и лейкоцитарная фракция отбиралась автоматической пипеткой в отдельную пробирку. После чего обе пробирки – с лейкоцитарной фракцией и остатком крови – замораживались и хранились в морозильнике при температуре –80 °С. Геномная ДНК выделялась при помощи High Pure PCR Template Preparation Kit (Roche) по протоколу производителя. После проверки качества ДНК и измерения ее концентрации на спектрофотометре NanoDrop 2000 от Thermo Scientific, образцы ДНК хранились до использования в морозильнике при –80 °С.
Для приготовления меченых зондов для гибридизации на микрочип использовали набор реагентов «BioPrime Total Genomic Labeling System» от Invitrogen, кат. № 18097-011. ДНК метили флуоресцентными красителями Alexa Fluor 3 или Alexa Fluor 5, которые аналогичны по свойствам широко используемым Cy3 и Cy5. Процедуру мечения проводили в соответствии с протоколом производителя.
Очистку меченых зондов от не включившейся метки проводили набором «BioPrime Purification Module» от Invitrogen, кат. № 46-6335. После очистки концентрацию меченой ДНК определяли на спектрофотометре NanoDrop 2000 от Thermo Scientific, и полученные зонды хранили при –20 °С до использования.
Перед гибридизацией меченых зондов на микрочипы последние обрабатывались в блокирующем растворе для «забивки» свободной поверхности стекла и снижения неспецифической светимости фона. Для этого использовали буфер – x3,5 SSC, 0,1 % SDS в 10 % растворе альбумина человека. Стекла с микрочипами выдерживали в этом буфере при температуре 30 °С в течение 1 часа. После чего промывали в дистиллированной воде 5 мин и высушивали на центрифуге.
Для гибридизации смешивали меченый зонд с буфером HybIt®Hybridization Solution от «Arrayit», кат. № HHS, прогревали 5 мин при 95 °С, быстро охлаждали на льду и наносили на микрочип, закрывали покровным стеклом и помещали в герметичной камере в термостат на 62 °С на 4–16 часов.
После гибридизации стекла с микрочипами промывали в отмывающем буфере А («Arrayit», кат. № WBA) при при 62 °С в течение 10 мин. Затем 10 мин при комнатной температуре в буфере В («Arrayit», кат. № WBВ), 5 минут при комнатной температуре в буфере С («Arrayit», кат. № WBС), ополаскивали водой и высушивали на центрифуге.
Микрочипы сканировали на сканере Agilent SureScan Microarray Scanner Bundle, кат. № G4900DA от «Agilent». Для анализа сканов микрочипов, оценки интенсивности сигналов и других характеристик микрочипов использовали программное обеспечение Feature Extraction Software License, кат. № G4461AA от «Agilent», адаптированное под формат созданного нами ДНК-микрочипа для выявления генетической предрасположенности к идиопатическим сколиозам. Интенсивность сигналов от «мутантного» и «нормального» вариантов каждого маркера представляли в виде графиков.
Графические результаты выборочно проверяли методом ПЦР. Для 10 маркеров были разработаны прямые аллель-специфичные праймеры и общий для каждой пары обратный праймер. Длина всех ПЦР продуктов была от 200 до 230 пар оснований. Фрагменты ДНК анализировались в 1 % агарозном геле и документировались на ChemiDoc MP Imaging System от Bio-Rad Laboratories, США.
Результаты исследования и их обсуждение
На рис. 1 в качестве примера приведен скан ДНК-микрочипа после гибридизации с меченым зондом из ДНК пациента под номером 259. Каждый олигонуклеотид напечатан в трех повторах. В нечетных рядах размещены «нормальные» аллели маркеров, под ними в четных рядах – «мутантные» аллели тех же маркеров. Разная величина точек обусловлена артефактами, возникшими при печати микрочипов. Величина пятна не связана с интенсивностью сигнала в нем.

Рис. 1. Скан результатов гибридизации меченого зонда из ДНК пациента на микрочип. В нечетных рядах напечатаны «нормальные» аллели маркеров, под ними, в четных рядах, напечатаны «мутантные» аллели тех же маркеров. Каждый из олигонуклеотидов напечатан в трех повторах
Различия в цвете определяются тем, что программа, формирующая сканы микрочипов, придает искусственный цвет пятнам в зависимости от разброса интенсивностей сигналов на микрочипе: черный – абсолютный минимум на отсканированном участке, белый – абсолютный максимум. Другие цвета на скане определяются отношением к этим двум крайним значениям.
Точки с очень слабым сигналом – отрицательные контроли, то есть фрагменты нечеловеческих (бактериальных и растительных) генов. Они помещены на микрочип потому, что эти последовательности ДНК не имеют аналогов в человеческом геноме. Существенный сигнал в этих точках говорит о неспецифической гибридизации и о том, что результатам такого эксперимента доверять не стоит. На микрочипе размещены и положительные контроли, это фрагменты генов «домашнего хозяйства» человека. Программа считывает сигнал из множества точек в каждом пятне и усредняет значения, затем числовые данные каждого скана переводятся в графическую форму. На рис. 2, показан график интенсивности сигналов от положительных и отрицательных контролей.
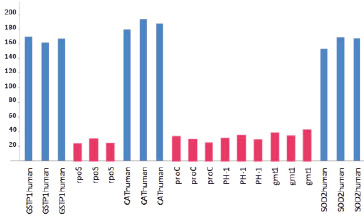
Рис. 2. График интенсивности сигналов от положительных (синий) и отрицательных (розовый) контролей
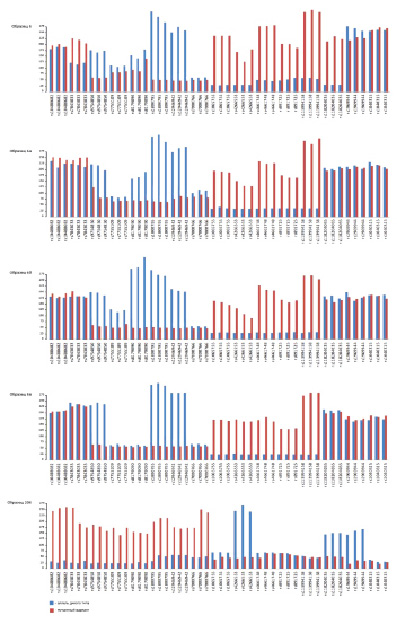
Рис. 3. Графики интенсивности сигналов от одинаковых маркеров в 5 образцах ДНК: образец № 6 – пациентка с диагностированным сколиозом; образец № 6а – ее брат; образец № 6б – ее мать; образец № 6в – сестра матери; образец № 206 – не родственная им пациентка
На рис. 3 показаны графики интенсивностей сигналов от «нормальных» и «мутантных» аллелей маркеров в различных образцах. Разница в интенсивности сигналов у разных маркеров, возможно, зависит от длины меченых фрагментов ДНК, которые связываются с напечатанными олигонуклеотидами.
Для мечения ДНК использовались случайные праймеры, которые неравномерно распределены по геному и в некоторых случаях могут формировать длинные последовательности, включившие больше флуоресцентной «метки». Концентрации всех напечатанных олигонуклеотидов выравнены, и различия в концентрациях составляют не более 1–2 процентов. Концентрации меченых зондов так же выравнивались, и на одну гибридизацию брали 10–12 нг ДНК. На графиках показаны одинаковые маркеры в 5 образцах и хорошо заметна разница в интенсивности сигналов от одинаковых маркеров у разных образцов. Образец № 6 – это пациентка с диагностированным сколиозом, образец № 6а – ее брат, образец № 6б – их мать, образец № 6в – сестра матери, образец № 206 – не родственная им пациентка. Хорошо заметен сходный профиль графиков у родственников и его отличие у стороннего человека. В некоторых случаях не удавалось однозначно установить статус носителя, то есть определить, является ли носитель гетерозиготой по конкретному маркеру или гомозиготой по одной из аллелей. Чтобы проверить результаты, полученные на микрочипах, мы использовали аллель-специфичную ПЦР.
На рисунке видно, что результаты анализа на микрочипах и методом ПЦР совпадают. В некоторых случаях интенсивность сигналов в трех повторах одного маркера достаточно сильно различаются. И хотя усредненные данные по аллелям позволяют установить статус носителя по данному маркеру, мы проверили некоторые такие случаи методом ПЦР. На рис. 5 показаны результаты такой проверки. Видно, что если интенсивности сигналов от двух пятен из трех близки, а сигнал от третьего пятна сильно отличается в ту или другую сторону, то результат анализа следует оценивать по интенсивности сигнала от двух пятен. Именно для таких случаев все маркеры на микрочипе напечатаны в трех повторах.
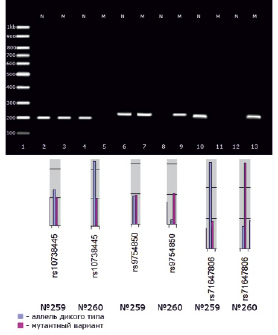
Рис. 4. Результаты верификации данных, полученных на микрочипах, методом аллель-специфичной ПЦР. Вверху рисунка литерами «N» и «M» обозначены дорожки на геле для «нормального» и «мутантного» вариантов маркеров. Под каждой парой дорожек помещены фрагменты графиков интенсивности сигналов от одинаковых маркеров у разных пациентов
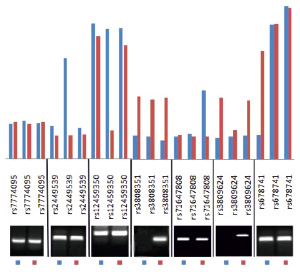
Рис. 5. Проверка результатов, полученных на микрочипах методом аллель-специфичной ПЦР. Показаны фрагменты графиков с интенсивностью сигналов от маркеров у разных пациентов. В некоторых случаях интенсивность сигнала одного из трех повторов каждого маркера сильно отличается от остальных двух повторов
На рис. 4 показаны результаты верификации результатов анализа на микрочипах методом аллель-специфичной ПЦР. Графики, из которых были взяты фрагменты на этом рисунке, построены по усредненным для трех точек данным.
Полученные результаты показывают, что разработанная тест-система для выявления генетической предрасположенности к идиопатическим сколиозам хорошо дискриминирует разные аллели маркеров, даже если они отличаются заменой только одного нуклеотида. Таким образом, чувствительность тест-системы вполне отвечает тем целям, которые были поставлены при ее разработке. Тест-система была запатентована как полезная модель [13].
Заключение
Созданный ДНК-микрочип позволяет надежно выявлять в геномах молекулярные маркеры предрасположенности к заболеванию идиопатическим сколиозом и маркеры прогрессирования этого заболевания. Так же он вполне надежно различает аллели маркеров, что позволяет определять генотипы пациентов. Такая информация позволит провести поиск ассоциаций между маркерами и клиническими формами идиопатического сколиоза. В дальнейшем, знание генетического статуса индивида (пациента) позволит принимать профилактические меры для предотвращения заболевания или снижения его рисков, а также выбирать стратегию лечения сколиоза с учетом генетики пациента. То есть созданный ДНК-микрочип даст возможность персонализированного подхода к лечению идиопатического сколиоза.
Библиографическая ссылка
Кузнецов С.Б., Михайловский М.В. СОЗДАНИЕ ДНК-МИКРОЧИПА ДЛЯ ВЫЯВЛЕНИЯ ГЕНЕТИЧЕСКОЙ ПРЕДРАСПОЛОЖЕННОСТИ К ИДИОПАТИЧЕСКОМУ СКОЛИОЗУ // Международный журнал прикладных и фундаментальных исследований. 2018. № 11-1. С. 67-74;URL: https://applied-research.ru/ru/article/view?id=12451 (дата обращения: 07.01.2026).

