Извлечение ценных компонентов из минерального сырья с помощью микроорганизмов служит признанным биотехнологическим методом, сочетающим в себе ряд преимуществ над традиционными способами. В эксплуатации микробного материала в бактериально-химических процессах переработки сульфидных руд (биовыщелачивание, биоокисление) существует два различных подхода. Первый включает применение эффективного сообщества, которое получают в результате продолжительного культивирования и адаптации. Первичный скрининг ацидофильных хемолитотрофных микроорганизмов проводят в колбах или лабораторных реакторах. Сообществами, пригодными к процессу, считают микробные консорции, стабильно окисляющие минералы и сохраняющие свой состав. Второй подход предполагает использование принудительно сформированного сообщества микроорганизмов, в которое входят по крайней мере один железо- и один сероокислитель. Дополнительно в него вводят гетеротрофные и/или миксотрофные организмы с целью утилизации метаболитов ключевых участников процесса. Как правило, они также способны к окислению двухвалентного железа и восстановленных соединений серы, тем самым обеспечивают растворение ценных металлов. Таким образом, формируют эффективную комбинацию микроорганизмов, состоящую из 2–4 видов [1, 2].
Применение микроорганизмов в биовыщелачивании уже продолжительное время ориентируется на смешанные и стабильно окисляющие культуры в противоположность ранее практикуемым при изучении кинетики процессов монокультурам. Из табл. 1 с литературными данными о качественном составе культур, используемых в бактериально-химических процессах переработки различных руд и концентратов, видно, что роды и виды консорциумов не отличаются богатым разнообразием и приблизительно похожи между собой (присутствуют железоокислитель и сероокислитель). Основными участниками являются мезофильные, умеренно и экстремально термофильные железо- и сероокисляющие бактерии и археи родов Acidithiobacillus, Leptospirillum, Sulfobacillus, Ferroplasma, Acidiphilium, Acidianus, Metallosphaera, Sulfolobus. Несмотря на общую скудность микроорганизмов, можно наблюдать различия в комбинациях их применения. Необходимо также учитывать факт нестерильности проведения бактериально-химических процессов, который может приводить к изменению качественного состава микробных ассоциаций.
Вопросы идентификации микроорганизмов с помощью молекулярно-биологических методов актуальны в области рудной микробиологии, поскольку позволяют оперативно и более информативно определять биоразнообразие ацидофильных хемолитотрофов в ходе биовыщелачивающих процессов.
Таким образом, состав микробного компонента следует рассматривать как один из факторов, определяющих успешность протекания процесса и стабильность кинетических показателей окисления для различных руд или концентратов.
В Научно-исследовательском геотехнологическом центре Дальневосточного отделения Российской академии наук (Камчатский край) проводятся исследования бактериально-химических процессов переработки сульфидной кобальт-медно-никелевой руды месторождения Шануч в периодическом и непрерывном режимах [20, 21]. В качестве микробного компонента в работах используется культура аборигенных ацидофильных хемолитотрофных микроорганизмов, выделенная из руды месторождения Шануч и культивируемая в лабораторных условиях, качественный состав которой подробно так и не был изучен.
Цель исследования заключалась в определении таксономического состава культуры микроорганизмов, ранее выделенной из сульфидной кобальт-медно-никелевой руды месторождения Шануч и используемой в лабораторных экспериментах по бактериально-химическому выщелачиванию, с применением высокопроизводительного секвенирования (метагеномного секвенирования 16S рРНК).
Материалы и методы исследования
Культура микроорганизмов и условия ее выделения. Исследуемая культура ацидофильных хемолитотрофных микроорганизмов была выделена из предварительно измельченного образца окисленной руды сульфидного кобальт-медно-никелевого месторождения Шануч (отвальный участок с интенсивными окислительными процессами). Накопление производили стационарно в колбе Эрленмейера объемом 500 мл при температуре 28 °С в термостате. В качестве питательной среды использовали 300 мл раствора минеральных солей Сильвермана и Люндгрена (9К) без FeSO4•7H2O. Масса образца руды составляла 25 г. Соотношение Т:Ж – 1:12. Культуру подвергали адаптации [20] и поддерживали на сульфидной руде месторождения, которую использовали в экспериментальных работах по биовыщелачиванию.
Выделение ДНК, ПЦР-амплификация, высокопроизводительное секвенирование на Illumina MiSeq, биоинформатическая обработка данных. Выделение ДНК, приготовление ДНК-библиотек, секвенирование, а также биоинформатическая обработка данных были проведены в Институте клеточного и внутриклеточного симбиоза Уральского отделения Российской академии наук сотрудниками центра коллективного пользования «Персистенция микроорганизмов» (г. Оренбург, Россия); дальнейшая обработка и интерпретация результатов – в Научно-исследовательском геотехнологическом центре Дальневосточного отделения Российской академии наук.
Выделение тотальной ДНК проводили с помощью механической гомогенизации в сочетании с методом ферментативного лизиса [22]. Для этого к 50 мкл осадка биомассы анализируемого образца добавляли 200 мкл ТСБ (20 ммоль/л ЭДТА, 750 ммоль/л NaCl, 100 ммоль/л Трис-HCl, рН 8,0). Клеточный лизис производили путем внесения 50 мкл ТСБ с лизоцимом в концентрации 10 мг/мл, после чего пробы инкубировали в течение 60 мин при 37 °С. Дальнейшее разрушение клеток осуществляли путем добавления лизирующего матрикса (Lysing matrix E, MP Biomedicals, США) в объеме 1/4 от объема пробирок и гомогенизацией содержимого в гомогенизаторе TissueLyser LT (Qiagen, Германия) в течение 1 мин при частоте 50 Гц. В составе матрикса находились: кремниевые шарики 0,1 мм, керамические шарики 1,4 мм и 1 стеклянный шарик 4 мм. Полученный раствор прогревали при 95 °С в течение 10 мин на твердотельном термостате Термит (ДНК-технология, Россия). Разрушение плазматических мембран клеток и гидролиз пептидных связей осуществляли добавлением 50 мкл 10 % раствора додецилсульфата натрия (итоговая концентрация 1 %) и 2 мкл раствора протеиназы К (10 мг/мл). Полученный раствор инкубировали при 60 °С в течение 60 мин. С целью денатурации и отделения белков вносили фенол-хлороформную смесь (1:1) в равном объеме (300 мкл) и встряхивали вручную 5 мин. Далее, центрифугировали при 14500 об/мин на высокоскоростной мини-центрифуге Microspin 12 с ротором MSR-12 (Bio-San, Латвия) в течение 5 мин при комнатной температуре. В отобранный объем водной фазы (250–300 мкл) добавляли равный объем хлороформ-изоамилового спирта (24:1), встряхивали в течение 5 мин и центрифугировали при 14500 об/мин при комнатной температуре 5 мин. В отделенный центрифугат добавляли 40 мкл 10 М ацетата аммония (10:1) и 800 мкл ледяного спирта (–20 °С). После выдержки нуклеиновых кислот при отрицательных температурах их центрифугировали. Полученную массу промывали холодным 80 % этиловым спиртом и центрифугировали на центрифуге с охлаждением для пробирок типа эппендорф до 2 мл (Universal 320 R с ротором 1689-А, Hettich, Германия) при 14000 об/мин сначала в течение 30 мин, а затем 10 мин при 4 °С. Осадок подсушивали. К полученному осадку приливали 30 мкл MQ воды, перемешивали на вортексе FV-2400 и встряхивали трижды. Чистоту ДНК контролировали с помощью электрофореза в 1 % агарозном геле и фотометрии Quantus (Promega, США) с применением набора Quanti Fluor dsDNA (Promega, США).
Таблица 1
Качественный состав культур микроорганизмов, используемых в бактериально-химическом выщелачивании (чановое и реакторное биовыщелачивание)
|
№ п/п |
Состав культур микроорганизмов |
Субстрат для биовыщелачивания |
Литература |
|
1 |
Acidithiobacillus spp, Leptospirillum spp., Acidiphilium spp. |
Никельсодержащая сульфидная руда |
[3] |
|
2 |
Sulfobacillus thermotolerans, S. thermosulfidooxidans, Acidithiobacillus caldus, Leptospirillum ferriphilum |
Медьсодержащий сульфидный флотоконцентрат |
[4] |
|
3 |
Мезофильная культура (Acidithiobacillus caldus, Leptospirillum ferriphilum, Sulfobacillus spp., Ferroplasma spp.) и экстремально термофильная культура (Acidianus brierleyi, Metallosphaera sedula, Sulfolobus spp.) |
Никель- и медьсодержащий концентрат |
[5] |
|
4 |
Acidithiobacillus caldus, Leptospirillum spp., Sulfobacillus spp., Ferroplasma spp. |
Медьсодержащие полиметаллические концентраты |
[6] |
|
5 |
Acidithiobacillus ferrooxidans, A. thiooxidans, Leptospirillum ferrooxidans, L. ferriphilum |
Пирротиновые золотосодержащие концентраты (BIOX™) |
[7] |
|
6 |
Acidithiobacillus ferrooxidans, A. thiooxidans, Leptospirillum ferrooxidans |
Никельсодержащие сульфидные концентраты (BioNIC®) |
[7] |
|
7 |
Leptospirillum spp., Acidithiobacillus spp., Sulfobacillus spp. |
Кобальт- и железосодержащий пиритовый концентрат |
[8] |
|
8 |
Acidithiobacillus ferrooxidans, A. thiooxidans, Leptospirillum ferrooxidans |
Дымовая пыль медеплавильного завода Sarcheshmeh |
[9] |
|
9 |
Acidithiobacillus caldus, Leptospirillum ferriphilum, Sulfobacillus spp., Ferroplasma spp. |
Халькопиритовый концентрат |
[10] |
|
10 |
Acidithiobacillus ferrooxidans, A. thiooxidans, Leptospirillium ferroxidans, Acidiphilium spp. |
Низкосортная никельсодержащая сульфидная руда |
[11, 12, 13] |
|
11 |
Acidithiobacillus ferrooxidans |
Урановая руда |
[14, 15] |
|
12 |
Acidithiobacillus caldus, Leptospirillum ferriphilum, Sulfobacillus benefaciens, S. thermosulfidooxidans |
Медный концентрат сланцевой руды |
[16] |
|
13 |
Acidithiobacillus ferrooxidans, A. thiooxidans, Leptospirillum ferrooxidans |
Сульфидные руды, содержащие сидерит, халькопирит, галенит и кварц |
[17] |
|
14 |
Acidithiobacillus ferrooxidans, A. thiooxidans, Sulfobacillus thermosulfidooxidans (DSMZ, штамм 9293) |
Сульфидный концентрат, содержащий пентландит, пирротин и халькопирит |
[18] |
|
15 |
Acidithiobacillus ferrooxidans и умеренно термофильная железоокисляющая бактерия (штамм MLY) |
Сульфид цинка |
[19] |
ДНК-библиотеки для секвенирования были созданы по протоколу Illumina (http://support.illumina.com/documents/documentation/chemistry_documentation/16s/16s-metagenomic-library-prep-guide-15044223-b.pdf) с праймерами к вариабельному участку V3-V4 гена 16S рРНК S-D-Bact-0341-b-S-17 и S-D-Bact-0785-a-A-21 [23]. В ходе приготовления ДНК-библиотек использовали стерильную ПЦР-воду (Евроген, Россия), Q5® High-Fidelity DNA Polymerase (New England Biolabs, США), смесь dNTP (Синтол, Россия). ПЦР фрагментов ДНК проводили с помощью термоциклера для амплификации нуклеиновых кислот Т100 (Bio-Rad Laboratories, США) в режиме: 95 °С (3 мин), 25 циклов (95 °С – 30 с, 56 °С – 30 с, 72 °С – 30 с), 72 °С (5 мин).
Секвенирование производили с помощью системы для автоматического секвенирования марки MiSeq (Illumina, США). Для этого использовали набор реактивов MiSeq Reagent Kit V3 для парно-концевого чтения 2×300 bp.
В ходе биоинформатической обработки производили оценку качества чтения ридов с помощью программы FastQC v0.11.5. На следующем этапе объединяли парно-концевые риды в контиги, используя программу PEAR. С помощью программы USEARCH 10.0.240_win32 осуществляли фильтрацию объединенных ридов по качеству и длине, используя команду fastq_filtr. Формирование операционных таксономических единиц (ОТЕ) проводили путем последовательной дерепликации и кластеризации на уровне 97 %. Для оценки количественной представленности полученных ОТЕ применяли глобальное выравнивание последовательностей ОТЕ на первоначальные объединенные риды. Из полученных данных исключали последовательности, встречающиеся однократно (синглтоны) или дважды (даблтоны). Таксономическую классификацию ОТЕ проводили с использованием базы данных RDP. Обилие и разнообразие микроорганизмов оценивали по количеству выделенных ОТЕ, индексам Chao1, Маргалефа, Шеннона и Симпсона.
Результаты исследования и их обсуждение
На этапе выделения нуклеиновых кислот из исследуемого образца культуры получили оптимальную ДНК для приготовления ДНК-библиотеки высокого качества.
В результате секвенирования было получено 18600 последовательностей фрагментов генов 16S рРНК, включающие 5580000 пар оснований. Средняя длина каждого фрагмента составляла 300 н.п. После последовательной дерепликации и кластеризации, удаления химерных последовательностей, глобального выравнивания последовательностей ОТЕ на первоначальные объединенные риды, удаления синглтонов и даблтонов риды были кластеризованы в 4 ОТЕ с 95 % сходством последовательностей.
Анализ значений операционных таксономических единиц показал, что выделенная культура, используемая в различных экспериментальных работах по биовыщелачиванию, характеризовалась маленьким количеством ОТЕ, что ожидаемо для полученных и культивируемых в лабораторных условиях ассоциаций микроорганизмов. Максимальное таксономическое богатство, оцениваемое по индексу Chao1, составляло 9. Оценка индексов Маргалефа, Шеннона и Симпсона показала, что разнообразие было незначительным в сравнении, например, с полученными ранее результатами [24] по определению таксономического состава накопительных культур из различных руд месторождения Шануч (табл. 2). Это указывает на факт сформированного состава биовыщелачивающего консорциума.
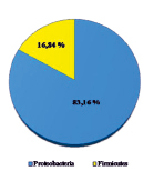
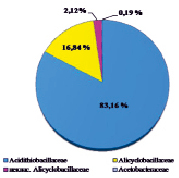
Оценивая микробный состав на основе данных с платформы Illumina, важным фактом являлось отсутствие в культуре архей на уровне домена; разнообразие было представлено исключительно бактериями. Структура культуры на уровнях филов, классов, семейств и родов приведена на рисунке.
В таксономическом отношении разнообразие было представлено бактериями филумов Proteobacteria и Firmicutes. Анализ обнаружил, что в составе микробной ассоциации имеются 3 класса (Alphaproteobacteria, Gammaproteobacteria, Bacilli), 4 семейства, включая 1 неклассифицированное (Acidithiobacillaceae, Alicyclobacillaceae, Acetobacteraceae, неклассиф. Alicyclobacillaceae), 4 рода, в том числе некультивированные (Acidithiobacillus spp., Acidiphilium spp., некульт. Alicyclobacillus spp., некульт. бактерия Firmicutes). До вида в культуре удалось определить ацидофильную железо- и сероокисляющую бактерию Acidithiobacillus ferrooxidans, являющуюся ключевым участником в бактериально-химических процессах окисления сульфидных руд.
Таблица 2
Индексы обилия и разнообразия культур микроорганизмов, выделенных из сульфидной руды кобальт-медно-никелевого месторождения Шануч
|
Культура |
Chao1 |
Маргалефа |
Шеннона |
Симпсона |
Литература |
|
Исследуемая культура |
9 |
0,34 |
0,23 |
0,71 |
– |
|
5 |
13,1 |
– |
1,01 |
0,39 |
[24] |
|
6 |
23 |
– |
0,49 |
0,80 |
|
|
35 |
7,13 |
– |
0 |
1,00 |
|
|
36 |
12,13 |
– |
1,27 |
0,33 |
|
|
49 |
9,25 |
– |
0,59 |
0,64 |
 а)
а) 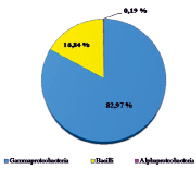 б)
б)
 в)
в) 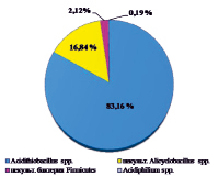 г)
г)
Структура бактериальной культуры, выделенной из сульфидной кобальт-медно-никелевой руды месторождения Шануч и используемой в лабораторных экспериментах по бактериально-химическому выщелачиванию, на уровне филов (a), классов (б), семейств (в) и родов (г)
Заключение
Определение качественного и количественного составов сообществ ацидофильных хемолитотрофных микроорганизмов, наряду с их активностью, – неотъемлемая часть исследований, касающихся бактериально-химического выщелачивания. Очевидно, что информация о видовом составе микрофлоры важна для обеспечения эффективности процесса.
В настоящей работе на основании результатов метагеномного секвенирования 16S рРНК изучен таксономический состав культуры, выделенной из сульфидной кобальт-медно-никелевой руды месторождения Шануч и используемой в лабораторных экспериментах по бактериально-химическому выщелачиванию одноименного месторождения. Показано присутствие ацидофильных железо- и сероокисляющих бактерий Acidithiobacillus ferrooxidans, Acidiphilium spp., а также некультивированных микроорганизмов.
Библиографическая ссылка
Хайнасова Т.С., Пашкевич Р.И. ТАКСОНОМИЧЕСКИЙ АНАЛИЗ КУЛЬТУРЫ АЦИДОФИЛЬНЫХ ХЕМОЛИТОТРОФНЫХ МИКРООРГАНИЗМОВ, ПРИНИМАЮЩЕЙ УЧАСТИЕ В БИОВЫЩЕЛАЧИВАНИИ СУЛЬФИДНОЙ РУДЫ МЕСТОРОЖДЕНИЯ ШАНУЧ // Международный журнал прикладных и фундаментальных исследований. 2019. № 10-1. С. 28-33;URL: https://applied-research.ru/ru/article/view?id=12862 (дата обращения: 10.03.2026).
DOI: https://doi.org/10.17513/mjpfi.12862

